概要
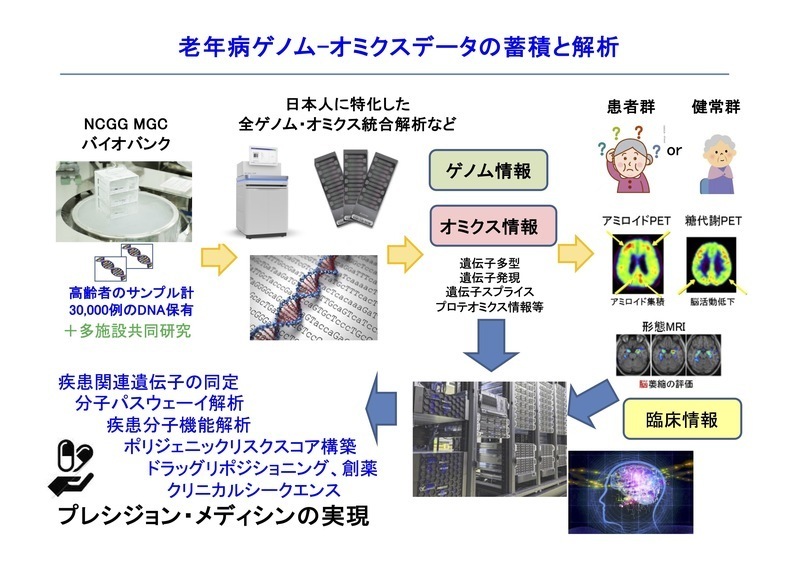
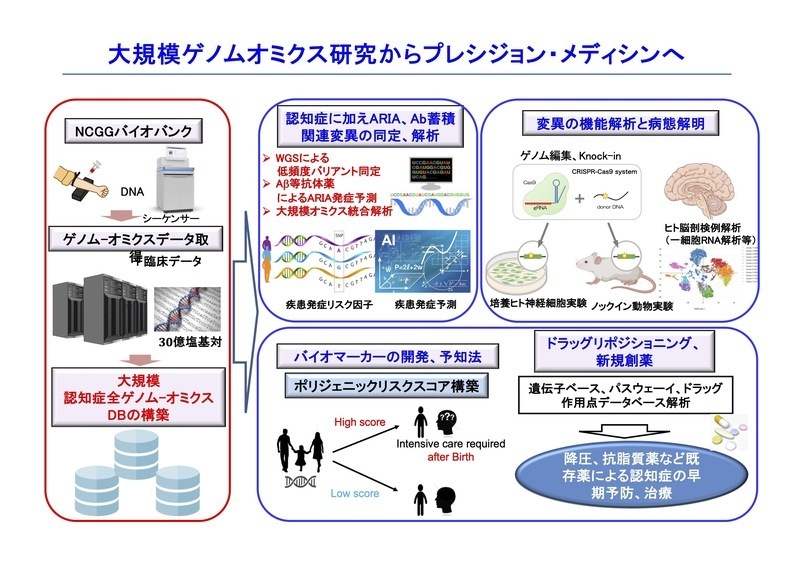
ヒト全ゲノム塩基配列が解読されて以来、網羅的なゲノム疫学、疾患ゲノム解析が可能になってきました。ゲノムは生命の設計図であり、その網羅的な解析を行えば病気などの表現型の全体像を見渡すことができるようになります。また、網羅的なゲノム解析研究は患者さんにご提供いただいたサンプルが原点であるため、解析の成果を直接、臨床の現場に還元できる可能性が大きいことや、研究の仮設を立てずに解析を進めるため、これまでには考えもしなかった疾患関連遺伝子やタンパク質を同定することが可能になり、疾患バイオマーカーや個々人に適した医療といった革新的な診断法や治療法の開発、すなわちプレシジョン・メディシンに繋がります。近年、このような網羅的ゲノム解析は、遺伝的多様性、遺伝子発現量の多様性といった、DNAやRNAの質的、量的な違いを知ることにより可能であることが明らかとなってきています。疾患ゲノム研究部では、主にNCGGバイオバンクで収集された検体について多様性からのアプローチを全ゲノム関連解析や次世代シークエンサー等を用いたゲノム解析手法および機械学習や人工知能を用いた情報科学等の高度な技術を駆使して推進します。また、家族性の認知症の一部には原因の遺伝子変異がわかっている症例もあり、遺伝子配列解析(クリニカルシーケンス)をすることで遺伝子診断にも貢献しています。このような解析を通じて老年病に関連した分子やそのネットワークの同定と解析を通したエビデンスに基づく予知予防法の開発や創薬シーズの探索につながる研究を積極的に進めています。
研究テーマ
-
網羅的ゲノム解析による老年病関連遺伝子の同定と解析
-
全ゲノム、エクソーム配列解析による老年病原因、関連遺伝子の同定と解析
-
全RNA配列解析とゲノム解析を統合した機能的疾患バリアントの同定と解析
-
ゲノム、オミクスデータを活用したドラッグリポジショニング
-
全ゲノム関連解析データを活用したポリジェニックリスクスコアの構築と活用
-
大規模ゲノム臨床情報統合データベースの構築と公開
-
大規模解析情報の機械学習や人工知能を用いた疾患発症予知法の開発および創薬関連シーズの探索
-
最新ゲノム、オミックス解析手法や情報解析法等の技術導入・開発

先頭へ戻る
メンバー
部長
尾崎浩一(Kouichi Ozaki)
E-mail: ozakikk(at)ncgg.go.jp
研究員
浅海裕也 (Yuya Asanomi)
光森理紗(Risa Mitsumori)
研究補助員
鈴木三惠(Mie Suzuki)
笹岡文(Fumi Sasaoka)
錦匠(Takumi Nishiki)
山根美奈(Mina Yamane)
深谷未奈(Mina Fukaya)
鈴木枝里子(Eriko Suzuki)
生田啓子(Keiko Ikuta)
金森久美子(Kumiko Kanamori)
先頭へ戻る
最近の原著論文
- A genome-wide association study identifies a novel East Asian-specific locus for dementia with Lewy bodies in Japanese subjects. Mitsumori R, Asanomi Y, Morizono T, Shigemizu D, Niida S, Ozaki K*. Molecular Medicine 31, 87 (2025).
doi: 10.1186/s10020-025-01115-7
- Alzheimer's disease may develop from changes in the immune system, cell cycle, and protein processing following alterations in ribosome function. Yamakawa A, Suganuma M, Mitsumori R, Niida S, Ozaki K, Shigemizu D. Scientific Reports 15, 3838 (2025).
doi: 10.1038/s41598-025-88526-y
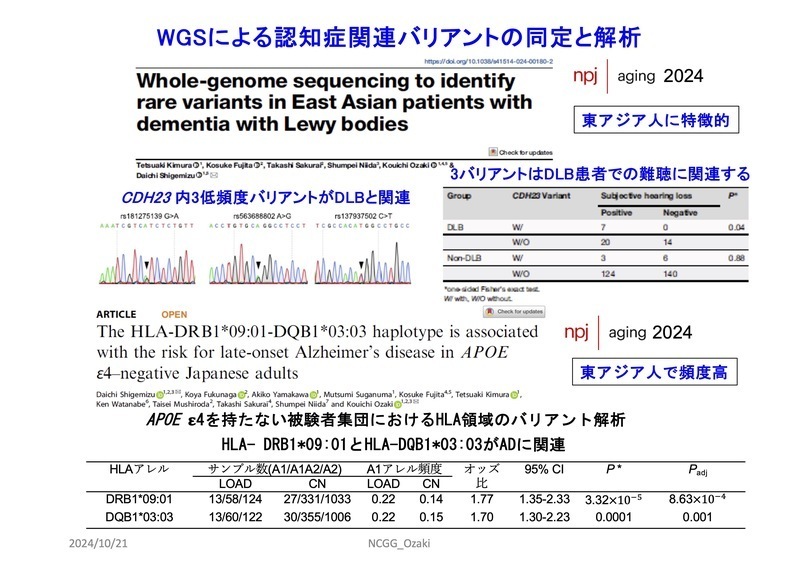
- Whole-genome sequencing to identify rare variants in East Asian patients with dementia with Lewy bodies. Kimura T, Fujita K, Sakurai T, Niida S, Ozaki K, Shigemizu D. NPJ Aging 10, 52 (2024).
doi: 10.1038/s41514-024-00180-2
- Population-specific non-coding and coding putative causal variants shape quantitative traits. Koyama S, Liu X, Koike Y, Hikino K, Koido M, Li W, Akaki K, Tomizuka K, Ito S, Otomo N, Suetsugu H, Yoshino S, Akiyama M, Saito K, Ishikawa Y, Benner C, Natarajan P, Ellinor PT, Mushiroda T, Horikoshi M, Ikeda1 M, Iwata N, Matsuda K, Biobank Japan Project, Niida S, Ozaki K, Momozawa Y, Ikegawa S, Takeuchi O, Ito K, Terao C. Nature Genetics 56(10), 2027-2035 (2024).
doi: 10.1038/s41588-024-01913-5
- A genome-wide association study identifies a locus associated with knee extension strength in older Japanese individuals. Ito S, Takuwa H, Kakehi S, Someya Y, Kaga H, Kumahashi N, Kuwata S, Wakatsuki T, Kadowaki M, Yamamoto S, Abe T, Takeda M, Ishikawa Y, Liu X, Otomo N, Suetsugu H, Koike Y, Hikino K, Tomizuka K, Momozawa Y, Ozaki K, Isomura M, Nabika T, Kaneko H, Ishijima M, Kawamori R, Watada H, Tamura Y, Uchio Y, Ikegawa S, Terao C. Communications Biology 7(1), 513 (2024).
doi: 10.1038/s42003-024-06108-6
- Molecular subtypes of lung adenocarcinoma present distinct immune tumor microenvironments. Fukuda H, Arai K, Mizuno H, Nishito Y, Motoi N, Arai Y, Hiraoka N, Shibata T, Sonobe Y, Kayukawa Y, Hashimoto E, Takahashi M, Fujii E, Maruyama T, Kuwabara K, Nishizawa T, Mizoguchi Y, Yoshida Y, Watanabe SI, Yamashita M, Kitano S, Sakamoto H, Nagata Y, Mitsumori R, Ozaki K, Niida S, Kanai Y, Hirayama A, Soga T, Tsukada K, Yabuki N, Shimada M, Kitazawa T, Natori O, Sawada N, Kato A, Yoshida T, Yasuda K, Ochiai A, Tsunoda H, Aoki K. Cancer Science 115(6), 1763-1777 (2024).
doi: 10.1111/cas.16154
- Identification of potential blood-based biomarkers for frailty by using an integrative approach. Suganuma M, Furutani M, Hosoyama T, Mitsumori R, Otsuka R, Takemura M, Matsui Y, Nakano Y, Niida S, Ozaki K, Satake S, Shigemizu D. Gerontology 70, 630-638 (2024).
doi: 10.1159/000538313
- Polygenic effects on the risk of Alzheimer’s disease in the Japanese population. Kikuchi M, Miyashita A, Hara N, Kasuga K, Saito Y, Murayama S, Kakita A, Akatsu H, Ozaki K, Niida S, Kuwano R, Iwatsubo T, Nakaya A, Ikeuchi T. Alzheimer's Research and Therapy 16(1), 45 (2024).
doi: 10.1186/s13195-024-01414-x
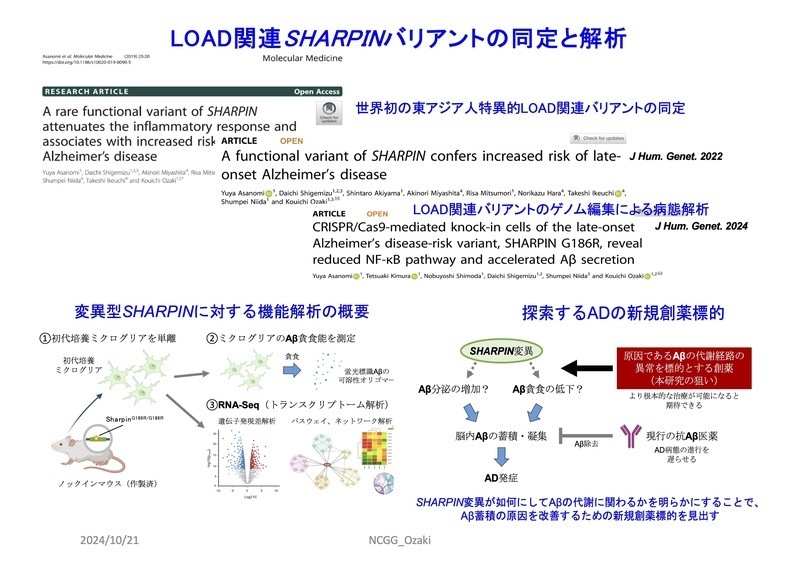
- CRISPR/Cas9-mediated knock-in cells of the late-onset Alzheimer’s disease-risk variant, SHARPIN G186R, reveal reduced NF-κB pathway and accelerated Aβ secretion. Asanomi Y, Kimura T, Shimoda N, Shigemizu D, Niida S, Ozaki K*. Journal of Human Genetics 69(5), 171-176 (2024).
doi: 10.1038/s10038-024-01224-x
- The HLA-DRB1*09:01-DQB1*03:03 haplotype is associated with the risk for late-onset Alzheimer’s disease in APOE ε4–negative Japanese adults. Shigemizu D, Fukunaga K, Yamakawa A, Suganuma M, Fujita K, Kimura T, Mushiroda T, Sakurai T, Niida S, Ozaki K*. npj Aging 10(1), 3 (2024).
doi: 10.1038/s41514-023-00131-3
- Exploring the genetic diversity of the Japanese population: Insights from a large-scale whole genome sequencing analysis. Kawai Y, Watanabe Y, Omae Y, Miyahara R, Khor SS, Noiri E, Kitajima K, Shimanuki H, Gatanaga H, Hata K, Hattori K, Iida A, Ishibashi-Ueda H, Kaname T, Kanto T, Matsumura R, Miyo K, Noguchi M, Ozaki K, et al. PLOS Genetics 19, e1010625 (2023).
doi: 10.1371/journal.pgen.1010625
- A functional variant SHARPIN confers increased risk of Alzheimer’s disease. Asanomi Y, Shigemizu D, Akiyama A, Mitsumori R, Miyashita A, Hara N, Ikeuchi T, Niida S, Ozaki K*. Journal of Human Genetics 67, 203-208 (2022).
doi: 10.1038/s10038-021-00987-x
- Multiancestry analysis of the HLA locus in Alzheimer’s and Parkinson’s diseases uncovers a shared adaptive immune response mediated by HLA-DRB1*04 subtypes. Guen YL, Luo G, Ambati A, Damotte V, . . . , Ozaki K, Sims R, Foo JN, Flier W, Ikeuchi T, Ramirez A, Mata I, Ruiz A, Gan-Or Z, Lambert JC, Greicius MD, Mignot E. Proceedings of the National Academy of Sciences of the United States of America 120(36), e2302720120 (2023).
doi: 10.1073/pnas.2302720120
- Trans-ethnic genomic informed risk assessment for Alzheimer's disease: An International Hundred K+ Cohorts Consortium study. Sleiman PM, Qu H, Connolly JJ, Mentch F, Pereira A, Lotufo PA, Tollman S, Choudhury A, Ramsay M, Kato N, Ozaki K, et al. on behalf of the DAC and IHCC consortium. Alzheimer's & Dementia 19(12), 5765-5772 (2023).
doi: 10.1002/alz.13378
- Genetic insights into ossification of the posterior longitudinal ligament of the spine. Koike Y, Takahata M, Nakajima M, Otomo N, Suetsugu H, Liu X, Endo T, Imagama S, Kobayashi K, Kaito T, Kato S, Kawaguchi Y, Kanayama M, Sakai H, Tsuji T, Miyamoto T, Inose H, Yoshii T, Kashii M, Nakashima H, Ando K, Taniguchi Y, Takeuchi K, Ito S, Tomizuka K, Hikino K, Iwasaki Y, Kamatani Y, Maeda S, Nakajima H, Mori K, Seichi A, Fujibayashi S, Kanchiku T, Watanabe K, Tanaka T, Kida K, Kobayashi S, Takahashi M, Yamada K, Takuwa H, Lu HF, Niida S, Ozaki K, et al. eLife 12, e86514 (2023).
doi: 10.7554/eLife.86514
- RNA-sequencing analysis identification of potential biomarkers for diagnosis of sarcopenia. Furutani M, Suganuma M, Akiyama S, Mitsumori R, Takemura M, Matsui Y, Satake S, Nakano Y, Niida S, Ozaki K, Hosoyama T, Shigemizu D. The journals of gerontology. Series A, Biological sciences and medical sciences 78(11), 1991-1998 (2023).
doi: 10.1093/gerona/glad150
- Classification and deep-learning-based prediction of Alzheimer disease subtypes by using genomic data. Shigemizu D, Akiyama S, Suganuma M, Furutani M, Yamakawa A, Nakano Y, Ozaki K, Niida S. Translational Psychiatry 13(1), 232 (2023).
doi: 10.1038/s41398-023-02531-1
- Tumor-infiltrating lymphocyte profiling defines three immune subtypes of NSCLC with distinct signaling pathways and genetic alterations. Nishito Y, Motoi N, Arai Y, Hiraoka N, Shibata T, Sonobe Y, Kayukawa Y, Hashimoto E, Takahashi M, Fujii E, Nishizawa T, Fukuda H, Ohashi K, Arai K, Mizoguchi Y, Yoshida Y, Watanabe S, Yamashita M, Kitano S, Sakamoto H, Nagata Y, Mitsumori R, Ozaki K, et al. Cancer Research Communications 3(6), 1026-1040 (2023).
doi: 10.1158/2767-9764.CRC-22-0415
- Cross-ancestry genome-wide analysis of atrial fibrillation provides new insights into disease biology and enables polygenic prediction of cardioembolic risk. Miyazawa K, Ito K, Ito M, Zou Z, Kubota M, Nomura S, Matsunaga H, Koyama S, Ieki H, Akiyama M, Kurosawa R, Yoshida H, Ozaki K, et al. Nature Genetics 55(2): 187-197 (2023).
doi: 10.1038/s41588-022-01284-9
- Deep learning-based age estimation from chest X-rays indicates cardiovascular prognosis. Ieki H, Ito K, Saji M, Kawakami R, Nagatomo Y, Takada K, Kariyasu T, Machida H, Koyama S, Yoshida H, Kurosawa R, Matsunaga H, Miyazawa K, Ozaki K, et al. Communications Medicine 2(1), 159 (2022).
doi: 10.1038/s43856-022-00220-6
- Identification of potential blood biomarkers for early diagnosis of Alzheimer’s disease through immune landscape analysis. Shigemizu D, Akiyama S, Mitsumori R, Niida S, Ozaki K*. npj Aging 8, 15 (2022).
doi: 10.1038/s41514-022-00096-9
- Whole-genome sequencing reveals novel ethnicity-specific rare variants associated with Alzheimer’s disease. Shigemizu D, Asanomi Y, Akiyama S, Mitsumori R, Niida S, Ozaki K*. Molecular Psychiatry 27, 2554-2562 (2022).
doi: 10.1038/s41380-022-01483-0
- Ethnic and trans-ethnic genome-wide association studies identify new loci influencing Japanese Alzheimer’s disease risk. Shigemizu D, Mitsumori R, Akiyama S, Miyashita A, Morizono T, Higaki S, Asanomi Y, Hara N, Tamiya G, Kinoshita K, Ikeuchi T, Niida S, Ozaki K*. Translational Psychiatry 11, 151 (2021).
doi: 10.1038/s41398-021-01272-3
以前の主要論文、「*」は責任著者、「†」は第一著者と同等
- Population-specific and trans-ancestry genome-wide analyses identify distinct and shared genetic risk loci for coronary artery disease. Koyama S, Ito K, Terao C, Akiyama M, Horikoshi M, Momozawa Y, Matsunaga H, Ieki H, Ozaki K, et al. Nature Genetics 52, 1169-1177 (2020).
doi: 10.1038/s41588-020-0705-3
- Large-scale genome-wide association study in a Japanese population identifies novel susceptibility loci across different diseases. Ishigaki K, Akiyama M, Kanai M, Takahashi A, Kawakami E, Sugishita H, Sakaue S, Matoba N, Low SK, Okada Y, Terao C, Amariuta T, Gazal S, Kochi Y, Horikoshi M, Suzuki K, Ito K, Koyama S, Ozaki K, et al. Nature Genetics 52(7), 669-679 (2020).
doi: 10.1038/s41588-020-0640-3
- A rare functional variant of SHARPIN attenuates the inflammatory response and associates with increased risk of late-onset Alzheimer's disease. Asanomi Y, Shigemizu D, Miyashita A, Mitsumori R, Mori T, Hara K, Ito K, Niida S, Ikeuchi T, Ozaki K*. Molecular Medicine 25(1), 20 (2019).
doi: 10.1186/s10020-019-0090-5
- Identification of six novel genetic loci associated with atrial fibrillation in Japanese population. Low SK, Takahashi A, Ebana Y, Ozaki K, et al. Nature Genetics 49, 953-958 (2017).
doi: 10.1038/ng.3842
- A functional SNP in FLT1 increases risk of coronary artery disease in a Japanese population. Konta A, Ozaki K*, et al. Journal of Human Genetics 61(5), 435-441 (2016).
doi: 10.1038/jhg.2015.171
- Molecular genetics of coronary artery diseases. Ozaki K*, Tanaka T. Journal of Human Genetics 61, 71-77 (2016).
doi: 10.1038/jhg.2015.70
- Genome-wide association study of peripheral arterial disease in a Japanese population. Matsukura M, Ozaki K, et al. PLoS ONE 10(10), e0139262 (2015).
doi: 10.1371/journal.pone.0139262
- Integrating genetic, transcriptional, and functional analyses to identify 5 novel genes for atrial fibrillation. Sinner MF, Tucker NR, Lunetta KL, Ozaki K†, et al. Circulation 130(15), 1225-1235 (2014).
doi: 10.1161/circulationaha.114.009892
- Novel genetic markers associate with atrial fibrillation risk in Europeans and Japanese. Lubitz SA, Lunetta KL, Lin H, Arking DE, Trompet S, Li G, Krijthe BP, Chasman DI, Barnard J, Kleber ME, Dörr M, Ozaki K†, et al. Journal of the American College of Cardiology 63(12), 1200-1211 (2014).
doi: 10.1016/j.jacc.2013.12.015
- Meta-analysis identifies six new susceptibility loci for atrial fibrillation. Ellinor PT, Lunetta KL, Albert CM, Glazer NL, Ritchie MD, Smith AV, Arking DE, Müller-Nurasyid M, Krijthe BP, Lubitz SA, Bis JC, Chung MK, Ozaki K†, et al. Nature Genetics 44, 670-675 (2012).
doi: 10.1038/ng.2261
- Genome-wide association study identified four novel susceptibility loci for Kawasaki disease. Onouchi Y, Ozaki K, et al. Nature Genetics 44, 517-521 (2012).
doi: 10.1038/ng.2220
- Common variants in CASP3 confer susceptibility to Kawasaki disease. Onouchi Y, Ozaki K, et al. Human Molecular Genetics 19(14), 2898-2906 (2010).
doi: 10.1093/hmg/ddq176
- SNPs in BRAP associated with risk of myocardial infarction in Asian populations. Ozaki K, et al. Nature Genetics 41, 329-333 (2009).
doi: 10.1038/ng.326
- A functional SNP in PSMA6 confers risk of myocardial infarction in the Japanese population. Ozaki K, et al. Nature Genetics 38, 921-925 (2006).
doi: 10.1038/ng1846
- Functional variation in LGALS2 confers risk of myocardial infarction and regulates lymphotoxin-α secretion in vitro. Ozaki K, et al. Nature 429, 72-75 (2004).
doi: 10.1038/nature02502
- Functional SNPs in the lymphotoxin-α gene that are associated with susceptibility to myocardial infarction. Ozaki K, et al. Nature Genetics 32, 650-654 (2002).
doi: 10.1038/ng1047
先頭へ戻る
主な講演、シンポジウム等(2019年以降)
-
Ozaki K. "Large-scale Genomic Research for Dementia"
The 11th NCGG – ICAH Symposium
2025年4月10日 大府
-
尾崎浩一 「大規模ゲノム解析を基盤とした老年病研究」
広島大学大学院セミナー
2024年10月7日 Web開催 招待講演
-
尾崎浩一 「大規模ゲノム解析を基盤とした老年病研究」
熊本大学大学院生命科学研究部附属健康長寿代謝制御研究センター・国立長寿医療研究センター 第2回共同シンポジウム
2024年3月30日 大府
-
尾崎浩一 「大規模ゲノム解析を基盤とした老年病研究」
第二回堺市認知症セミナー
2024年2月3日 大阪府 招待講演
-
尾崎浩一 「ゲノム解析を基盤とした老年病研究」
予測ゲノミクスオンラインセミナー2023
2023年10月24日 Web開催 招待講演
-
尾崎浩一 「大規模ゲノム解析を基盤とした老年病研究」
広島大学大学院セミナー
2023年10月30日 Web開催 招待講演
- 尾崎浩一 「老年病におけるゲノム医科学の現状と近未来」
広島大学大学院医系科学研究科 循環器内科学リサーチセミナー
2022年8月8日 WEB開催
- 尾崎浩一 「ゲノム医科学概論」
広島大学大学院医系科学研究科 3年次講義
2022年5月18日 WEB開催
- 尾崎浩一 「Large-scale genomic analysis for Alzheimer’s disease in Japanese」
第40回日本認知症学会学術集会 シンポジウム16
2021年11月28日 東京
- 尾崎浩一 「日本人における認知症のゲノム解析」
日本人類遺伝学会第66回大会 シンポジウム1 生活習慣病のゲノム研究の最前線
2021年10月14日 横浜
- 尾崎浩一 「ゲノム医科学概論」
広島大学大学院医系科学研究科 3年次講義
2021年6月7日 WEB開催
- Ozaki K. "Molecular genetics of cognitive impairment in Japanese"
Asian Forum of Alzheimer’s Disease (AFAD) 2019
2020年2月8日 東京
- 尾崎浩一 「NCGGにおける認知症のゲノム解析」
東京都健康長寿医療センター 教育セミナー
2020年1月14日 東京
- Ozaki K. "Molecular Genetics of Late-onset Alzheimer’s disease"
The 5th NCGG – ICAH Symposium
2019年4月11日 大府
先頭へ戻る























































