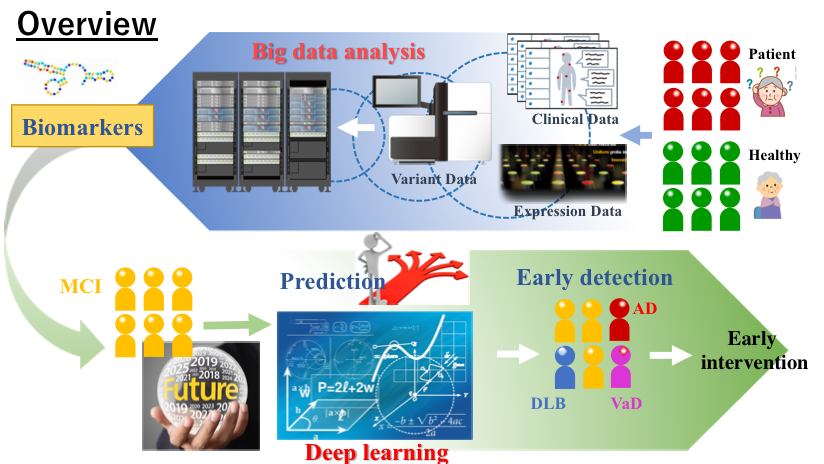
研究テーマ
近年、ヒト全ゲノム塩基配列や網羅的な遺伝子発現やその遺伝子発現を制御するマイクロRNA (miRNA) をはじめとした大量のヒトゲノムデータ解析が可能になってきました。この大量の網羅的なデータから、遺伝統計解析手法により疾患バイオマーカーの同定や個人に適した医療といった革新的な診断法や治療法の開発、すなわちプレシジョン・メディシンの研究を行なっています。
1. 疾患原因変異同定法の開発
- 次世代シークエンスデータから高精度なバリアントの同定
- 次世代シークエンスデータから中間サイズインデル(挿入・欠失)の同定
2. ゲノム・オミックス、臨床情報を用いた統合解析による疾患原因遺伝子の探索
3. 機械学習・人工知能による早期予防を目的とした疾患発症予測モデルの開発
- バリアントデータから疾患発症リスク予測
- 遺伝子発現情報から生存率・再発率予測
- バリアントデータと発現データの統合解析による早期認知症発症リスク予測
先頭へ戻る
メンバー
部長
重水 大智 (Daichi Shigemizu), Ph.D
専門: 遺伝統計学, バイオインフォマティクス, 次世代シークエンス解析, GWAS解析
E-mail: daichi (at) ncgg.go.jp
研究員
- 木村 哲晃 (Tetsuaki Kimura), Ph.D
外来研究員
- 古谷 元樹 (Motoki Furutani), Ph.D
研究補助者
先頭へ戻る
業績
発表論文 (*: 責任著者)
2025年
- Sakai T., Furutani M., Nakashima M., Ishibashi N., Maeda J., Oguri N., Miyamoto S., Miyauchi S., Okamura S., Okubo Y., Tokuyama T., Oda N., Mitsumori R., Niida S., Ozaki K., Shigemizu D.*, and Nakano Y.*. Genome-wide association study of atrial fibrillation recurrence after radiofrequency catheter ablation in a Japanese population. J Cardiovasc Electrophysiol. 2025. (accepted)
- Furutani M., Kimura T., Fukunaga K., Suganuma M., Takemura M., Matsui Y., Satake S., Nakano Y., Mushiroda T., Niida S., Ozaki K., Hosoyama T.*, and Shigemizu D*. Identification of a risk allele at SLC41A3 and a protective allele HLA-DPB1*02:01 associated with sarcopenia in Japanese. Gerontology. 2025. accepted (News & Topics)
- Mitsumori R., Asanomi Y., Morizono T., Shigemizu D., Niida S., and Ozaki K. A genome-wide association study identifies a novel East Asian-specific locus for dementia with Lewy bodies in Japanese subjects.
 Mol Med. 2025 Mar 6;31(1):87. (プレスリリース)
Mol Med. 2025 Mar 6;31(1):87. (プレスリリース)
- Yamakawa A., Suganuma M., Mitsumori R., Niida S., Ozaki K., and Shigemizu D.* Alzheimer’s disease may develop from changes in the immune system, cell cycle, and protein processing following alterations in ribosome function
 . Sci Rep. 2025 Jan 30;15(1):3838. (News & Topics)
. Sci Rep. 2025 Jan 30;15(1):3838. (News & Topics)
2024年
- Kimura T., Fujita K., Sakurai T., Niida S., Ozaki K., and Shigemizu D.* Whole-genome sequencing to identify rare variants in East Asian patients with dementia with Lewy bodies
 . NPJ Aging. 2024 Nov 21;10(1):52. (News & Topics)
. NPJ Aging. 2024 Nov 21;10(1):52. (News & Topics)
- Suganuma M., Furutani M., Hosoyama T., Mitsumori R., Otsuka R., Takemura M., Matsui Y., Nakano Y., Niida S., Ozaki K., Satake S., and Shigemizu D.* Identification of potential blood-based biomarkers for frailty by using an integrative approach
 . Gerontology. 2024;70(6):630-638. (News & Topics)
. Gerontology. 2024;70(6):630-638. (News & Topics)
- Asanomi Y., Kimura T., Shimoda N., Shigemizu D., Niida S., and Ozaki K. CRISPR/Cas9-mediated knock-in cells of the late-onset Alzheimer’s disease-risk variant, SHARPIN G186R, reveal reduced NF-κB pathway and accelerated Aβ secretion
 . J Hum Genet. 2024 May;69(5):171-176. (News & Topics)
. J Hum Genet. 2024 May;69(5):171-176. (News & Topics)
- Shigemizu D.*, Fukunaga K., Yamakawa A., Suganuma M., Fujita K., Kimura T., Watanabe K., Mushiroda T., Sakurai T., Niida S., and Ozaki K. The HLA-DRB1*09:01-DQB1*03:03 haplotype is associated with the risk for late-onset Alzheimer’s disease in APOE ε4–negative Japanese adults
 . NPJ Aging. 2024 Jan 2;10(1):3. (プレスリリース)
. NPJ Aging. 2024 Jan 2;10(1):3. (プレスリリース)
- Li J., Hosoyama T., Shigemizu D., Yasuoka M., Kinoshita K., Maeda K., Takemura M., Matsui Y., Arai H., and Satake S.. Association between circulating levels of CXCL9 and CXCL10 and physical frailty in older adults
 . Gerontology. 2024;70(3):279-289.
. Gerontology. 2024;70(3):279-289.
2023年
-
Shigemizu D.*, Akiyama S., Suganuma M., Furutani M., Yamakawa A., Nakano Y., Ozaki K., and Niida S. Classification and deep-learning-based prediction of Alzheimer's disease subtypes by using genomic data. Transl Psychiatry. 2023 June 29;13:232.
Transl Psychiatry. 2023 June 29;13:232.
-
Furutani M., Suganuma M., Akiyama S., Mitsumori R., Takemura M., Matsui Y., Satake S., Nakano Y., Niida S., Ozaki K., Hosoyama T., and Shigemizu D.* RNA-sequencing analysis identification of potential biomarkers for diagnosis of sarcopenia. J Gerontol A Biol Sci Med Sci. 2023 Jun 22;glad150.
J Gerontol A Biol Sci Med Sci. 2023 Jun 22;glad150.
-
重水 大智: 認知症の遺伝について (2023). 認知症サポートSOMPO笑顔倶楽部.
-
重水 大智: バイオインフォマティクスの世界 (2023). 別冊 医学のあゆみ, pp.51-59.
2022年
- Shigemizu D.*, Akiyama S., Mitsumori R., Niida S., and Ozaki K. Identification of potential blood biomarkers for early diagnosis of Alzheimer’s disease through immune landscape analysis.
 NPJ Aging. 2022 Nov 4;8(1):15.
NPJ Aging. 2022 Nov 4;8(1):15.
- Shigemizu D.*, Asanomi Y., Akiyama S., Higaki S., Sakurai T., Ito K., Niida S., and Ozaki K. Network-based meta-analysis and the candidate gene association studies reveal novel ethnicity-specific variants in MFSD3 and MRPL43 assocated with dementia with Lewy bodies.
 Am J Med Genet B Neuropsychiatr Genet. 2022 Jun 28.
Am J Med Genet B Neuropsychiatr Genet. 2022 Jun 28.
- Shigemizu D.*, Asanomi Y., Akiyama S., Mitsumori R., Niida S., and Ozaki K. Whole-genome sequencing reveals novel ethnicity-specific rare variants associated with Alzheimer's disease.
 Mol Psychiatry. 2022 Mar 10.
Mol Psychiatry. 2022 Mar 10.
- 重水 大智: 疾患バイオマーカーとしてのマイクロRNAと診断応用 (2022). シーエムシー出版, 第Ⅰ編第22章 pp.176-181.
- 重水 大智: 疾患ゲノム解析II: GWAS解析 (2022). 医学のあゆみ, Vol.281, No.9, 920-923.
- 重水 大智: 疾患ゲノム解析I: NGS解析 (2022). 医学のあゆみ, Vol.281, No.8, 854-858.
2021年
- Asanomi Y., Shigemizu D., Akiyama S., Miyashita A., Mitsumori R., Hara N., Ikeuchi T., Niida S., and Ozaki K. A functional variant of SHARPIN confers increased risk of late-onset Alzheimer’s disease.
 J Hum Genet. 2021 Nov 5.
J Hum Genet. 2021 Nov 5.
- Akiyama S., Higaki S., Ochiya T., Ozaki K., Niida S.*, and Shigemizu D.* JAMIR-eQTL: Japanese genome–wide identification of microRNA expression quantitative trait loci across dementia types.
 Database (Oxford) 2021 Nov 3;2021(2021):baab072.
Database (Oxford) 2021 Nov 3;2021(2021):baab072.
- Asanomi Y., Shigemizu D.*, Akiyama S., Sakurai T., Ozaki K., Ochiya T., and Niida S*. Dementia subtype prediction models constructed by penalized regression methods for multiclass classification using serum microRNA expression data.
 Sci Rep. 2021 Oct 22;11(1):20947.
Sci Rep. 2021 Oct 22;11(1):20947.
- Fujimoto A, Wong JH, Yoshii Y, Akiyama S, Tanaka A, Yagi H, Shigemizu D, Nakagawa H, Mizokami M, Shimada M. Whole-genome sequencing with long reads reveals complex structure and origin of structural variation in human genetic variations and somatic mutations in cancer.
 Genome Med. 2021 Apr 29;13(1):65.
Genome Med. 2021 Apr 29;13(1):65.
- Shigemizu D.*, Mitsumori R., Akiyama S., Miyashita A., Morizono T., Higaki S., Asanomi Y., Hara N., Tamiya G., Kinoshita K., Ikeuchi T., Niida S., and Ozaki K. Ethnic and trans-ethnic genome-wide association studies identify new loci influencing Japanese Alzheimer’s disease risk.
 Transl Psychiatry.2021 Mar 3;11(1):151.
Transl Psychiatry.2021 Mar 3;11(1):151.
- 重水 大智, 新飯田 俊平: 認知症のゲノム解析 (2021). 医学のあゆみ, Vol.279, No.5, 22360-22364.
- 重水 大智, 新飯田 俊平: 血中マイクロRNAの現状 (2021). Geriat. Med., Vol.59, No.2, 163-167.
2020年
- Shigemizu D.*, Akiyama S., Higaki S., Sugimoto T., Sakurai T., Boroevich KA., Sharma A., Tsunoda T., Ochiya T., Niida S., and Ozaki K. Prognosis prediction model for conversion from mild cognitive impairment to Alzheimer’s disease created by integrative analysis of multi-omics data.
 Alzheimers Res Ther. 2020 Nov 10;12(1):145.
Alzheimers Res Ther. 2020 Nov 10;12(1):145.
- Mitsumori R., Sakaguchi K., Shigemizu D., Mori T., Akiyama S., Ozaki K., Niida S., and Shimoda N. Lower DNA methylation levels in CpG island shores of CR1, CLU, and PICALM in the blood of Japanese Alzheimer's disease patients.
 PLoS One. 2020 Sep 29;15(9):e0239196.
PLoS One. 2020 Sep 29;15(9):e0239196.
- Shigemizu D.*, Mori T., Akiyama S., Higaki S., Watanabe H., Sakurai T., Niida S., and Ozaki K. Identification of potential biomarkers for early diagnosis of Alzheimer's disease through RNA sequencing analysis.
 Alzheimers Res Ther. 2020 Jul 16;12(1):87.
Alzheimers Res Ther. 2020 Jul 16;12(1):87.
- 重水 大智, 新飯田 俊平: 血中マイクロRNAからの認知症予測 (2020). 医学のあゆみ, Vol.272, No.3, 244-245.
2019年
- Chandra A., Sharma A., Dehzangi A., Shigemizu D., and Tsunoda T. Bigram-PGK: phosphoglycerylation prediction using the technique of bigram probabilities of position specific scoring matrix.
 BMC Mol Cell Biol. 2019 Dec 20;20(Suppl 2):57.
BMC Mol Cell Biol. 2019 Dec 20;20(Suppl 2):57.
- Shigemizu D.*, Akiyama S., Asanomi Y., Boroevich KA., Sharma A., Tsunoda T., Sakurai T., Ozaki K., Ochiya T., and Niida S. A comparison of machine learning classifiers for dementia with Lewy bodies using miRNA expression data.
 BMC Med Genomics. 2019 Oct 30;12(1):150.
BMC Med Genomics. 2019 Oct 30;12(1):150.
- Sharma A., Vans E., Shigemizu D., Boroevich KA., and Tsunoda T. DeepInsight: a methodology to transform a non-image data to an image for convolution neural network architecture.
 Sci Rep. 2019 Aug 6;9(1):11399.
Sci Rep. 2019 Aug 6;9(1):11399.
- Wong JH., Shigemizu D., Yoshii Y., Akiyama S., Tanaka A., Nakagawa H., Narumiya S., and Fujimoto A. Identification of intermediate-sized deletions and inference of their impact on gene expression in a human population.
 Genome Med. 2019 Jul 24;11(1):44.
Genome Med. 2019 Jul 24;11(1):44.
- Asanomi Y., Shigemizu D., Miyashita A., Mitsumori R., Mori T., Ito K., Niida S., Ikeuchi T., and Ozaki K. A rare functional variant of SHARPIN attenuates the inflammatory response and associates with increased risk of late-onset Alzheimer's disease.
 Mol Med. 2019 Jun 20;25(1):2.
Mol Med. 2019 Jun 20;25(1):2.
- Singh V., Sharma A., Chandra A., Dehzangi A., Shigemizu D., and Tsunoda T. Computational prediction of Lysine pupylation sites in prokaryotic proteins using position specific scoring matrix into bigram for feature extraction. Lect Notes in Artificial Intelligence, 2019 Part III, pp.488-500.
- Vans E., Sharma A., Patil A., Shigemizu D., and Tsunoda T. Clustering of Small-Sample Single-Cell RNA-Seq data via Feature Clustering and Selection. Lect Notes in Artificial Intelligence, 2019 Part III, pp.445-456.
- Shigemizu D.*, Akiyama S., Asanomi Y., Boroevich KA, Sharma A., Tsunoda T., Matsukuma K., Ichikawa M., Sudo H., Takizawa S., Sakurai T., Ozaki K., Ochiya T., and Niida S. Risk prediction models for dementia constructed by supervised principal component analysis using miRNA expression data.
 Commun Biol. 2019 Feb.25;2:77.
Commun Biol. 2019 Feb.25;2:77.
- Reddy HM., Sharma A., Dehzangi A., Shigemizu D., Chandra A., and Tsunoda T. GlyStruct: Glycation prediction using structural properties of amino acid residues.
 BMC Bioinformatics. 2019 19(Suppl 13):547.
BMC Bioinformatics. 2019 19(Suppl 13):547.
- 重水 大智, 角田 達彦: 機械学習・AIを駆使した多因子疾患発症・進展の予測モデルの開発 (2019). 内分泌・糖尿病・代謝内科, 11月号, Vol.49, No.5, 363-366.
- 菊地 正隆, 宮下 哲典, 原 範和, 重水 大智, 尾崎 浩一, 新飯田 俊平, 池内 健, 中谷 明弘: 日本人集団におけるアルツハイマー病ポリジェニック解析 (2019). Dementia Japan, 10月号, Vol.33 (4) 562.
- 重水 大智, 新飯田 俊平: 血液から認知症診断技術 (2019). PHARMSTAGE, 9月号, Vol.19, No.6, 55-58.
2018年
- Yamaguchi-Kabata Y., Morihara T., Ohara T., Ninomiya T., Takahashi A., Akatsu H., Hashizume Y., Hayashi N., Shigemizu D., Boroevich KA., Ikeda M., Kubo M., Takeda M., and Tsunoda T. Integrated analysis of human genetic association study and mouse transcriptome suggests LBH and SHF genes as novel susceptible genes for amyloid-β accumulation in Alzheimer’s disease.
 Hum Genet. 2018 Jul. 137(6-7):521-533.
Hum Genet. 2018 Jul. 137(6-7):521-533.
- López Y., Kamola PJ., Sharma R., Shigemizu D., Tsunoda T., and Sharma A. Computational pipelines and workflows in Bioinformatics.
 Encyclopedia of Bioinformatics and Computational Biology 2018 Sep 1, 20089;1-24.
Encyclopedia of Bioinformatics and Computational Biology 2018 Sep 1, 20089;1-24.
- Nishino J., Kochi Y., Shigemizu D., Kato M., Ikari K., Ochi H., Noma H., Matsui K., Morizono T., Boroevich KA., Tsunoda T., and Matsui S. Empirical Bayes estimation of semi-parametric hierarchical mixture models for unbiasedcharacterization of polygenic disease architectures.
 Front Genet. 2018 Apr 24, 9:115.
Front Genet. 2018 Apr 24, 9:115.
- Shigemizu D.*, Miya F., Akiyama S., Okuda S., Boroevich KA., Fujimoto A., Nakagawa H., Ozaki K., Niida S., Kanemura Y., Okamoto N., Saitoh S., Kato M., Yamasaki M., Matsunaga T., Mutai H., Kosaki K., and Tsunoda T. IMSindel: An accurate intermediate-size indel detection tool incorporating de novo assembly and gapped global-local alignment with split read analysis.
 Sci Rep. 2018 Apr 4;8(1):5608.
Sci Rep. 2018 Apr 4;8(1):5608.
- 重水 大智: 乳がんのゲノム医療 - 生存期間・再発期間予後予測解析 (2018). 外科, 11月号, Vol.80, No.12, 1205-1209.
- 重水 大智: エクソーム解析による疾患ゲノム研究 (2018). 細胞, 5月号, Vol.50, No.5, 45-48.
2017年
- Shigemizu D., Iwase T., Yoshimoto M., Suzuki Y., Miya F., Boroevich KA., Katagiri T., Zembutsu H., and Tsunoda T. The prediction models for postoperative overall survival and disease-free survival in patients with breast cancer.
 Cancer Med. 2017 Jul;6(7):1627-1638.
Cancer Med. 2017 Jul;6(7):1627-1638.
- Sharma A., Boroevich KA., Shigemizu D., Kamatani Y., Kubo M., and Tsunoda T. Hierarchical Maximum Likelihood Clustering Approach.
 IEEE Trans Biomed Eng. 2017 Jan;64(1):112-122.
IEEE Trans Biomed Eng. 2017 Jan;64(1):112-122.
- 重水 大智: エクソームシークエンスとプレシジョン・メディシン (2017). アンチ・エイジング医学, 10月号, Vol.13, No.5, 36-40.
- 重水 大智: エクソーム解析によるこれからの疾患ゲノム研究 (2017). 細胞, 7月号, Vol.49, No.8, 48-51.
- 重水 大智: エクソーム解析によるこれからの疾患ゲノム研究 (2017). Bio Clinica, 6月号, Vol.32, No.6, 591-594.
2016年
- Ichikawa M., Aiba T., Ohno S., Shigemizu D., Ozawa J., Sonoda K., Fukuyama M., Itoh H., Miyamoto Y., Kubo M., Tsunoda T., Makiyama T., Tanaka T., Shimizu W., and Horie M. Phenotypic Variability of ANK2 Mutations in Patients with Inherited Primary Arrhythmia Syndromes.
 Circ J. 2016 Nov 25;80(12):2435-2442.
Circ J. 2016 Nov 25;80(12):2435-2442.
- Sharma A., Shigemizu D., Boroevich KA., Lopez Y., Kamatani Y., Kubo M., and Tsunoda T. Stepwise Iterative Maximum Likelihood Clustering Approach.
 BMC Bioinformatics. 2016 Aug 24;17(1):319.
BMC Bioinformatics. 2016 Aug 24;17(1):319.
- Yagihara N., Watanabe H., Barnett P., Duboscq-Bidot L., Thomas AC., Yang P., Ohno S., Hasegawa K., Kuwano R., Chate S., Redon R., Schott JJ., Probst V., Koopmann TT., Bezzina CR., Wilde AAM., Nakano Y., Aiba T., Miyamoto Y., Kamakura S., Darbar D., Donahue BS., Shigemizu D., Tanaka T., Tsunoda T., Suda M., Sato A., Minamino T., Endo N., Shimizu W., Horie M., Roden DM., and Makita N. Variants in the SCN5A promoter associated with various arrhythmia phenotypes.
 J Am Heart Assoc. 2016 Sep 13;5(9):e003644.
J Am Heart Assoc. 2016 Sep 13;5(9):e003644.
- Morishita M., Muramatsu T., Suto Y., Hirai M., Konishi T., Shigemizu D., Tsunoda T., Moriyama K., and Inazawa J. Chromothripsis-like chromosomal rearrangements induced byionizing radiation using proton microbeam irradiation system.
 Oncotarget. 2016 Mar 1;7(9):10182-92.
Oncotarget. 2016 Mar 1;7(9):10182-92.
- 重水 大智,桃沢 幸秀,久保 充明,角田 達彦: 目的にあったエクソーム濃縮キットの選択ポイント (2016). 実験医学, 4月号, Vol.34, No.6, 955-960.
2015年
- Aiba T., Shigemizu D., Nakagawa H., Ozaki K., Miya F., Satake W., Toda T., Miyamoto Y., Fujimoto A., Suzuki Y., Kubo M., Tsunoda T., Kusano K., Yasuda S., Ogawa H., Tanaka T. and Shimizu W. Calmodulin interacting genes as a novel candidate for pathogenesis of long-QT syndrome.
 Circulation. 2015 132(Suppl 3) A12257.
Circulation. 2015 132(Suppl 3) A12257.
- Shigemizu D., Momozawa Y., Abe T., Morizono T., Boroevich KA., Takata S., Ashikawa K., Kubo M., and Tsunoda T. Performance of comparison of four commercial human whole-exome capture platforms.
 Sci Rep. 2015 Aug 3;5:12742.
Sci Rep. 2015 Aug 3;5:12742.
- Shigemizu D., Aiba T., Nakagawa H., Ozaki K., Miya F., Satake W., Toda T., Miyamoto Y., Fujimoto A., Suzuki Y., Kubo M., Tsunoda T., Shimizu W., and Tanaka T. Exome analyses of long QT syndrome reveal candidate pathogenic mutations in calmodulin-interacting gene.
 PLoS One. 2015 10(7), e0130329.
PLoS One. 2015 10(7), e0130329.
- Miya F., Kato M., Shiohama T., Okamoto N., Saitoh S., Yamasaki M., Shigemizu D., Abe T., Morizono T., Boroevich KA., Kosaki K., Kanemura Y., and Tsunoda T. A combination of targeted enrichment methodologies for whole-exome sequencing reveals novel pathogenic mutations.
 Sci Rep. 2015 Mar 19;5:9331.
Sci Rep. 2015 Mar 19;5:9331.
- Fujimoto A., Furuta M., Shiraishi Y., Gotoh K., Kawakami Y., Arihiko K., Nakamura T., Ueno M., Ariizumi S., Nguyen H., Shigemizu D., Abe T., Boroevich KA., Nakano K., Sasaki A., Kitada R., Maejima K., Yamamoto Y., Tanaka H., Shibuya T., Shibata T., Ojima H., Shimada K., Hayami S., Shigekawa Y., Aikata H., Ohdan H., Marubashi S., Yamada T., Kubo M., Hirano S., Ishikawa O., Yamamoto M., Yamaue H., Yamaue H., Chayama K., Miyano S., Tsunoda T., and Nakagawa H. Whole-genome mutational landscape of liver cancers displaying biliary phenotype reveals hepatitis impact and molecular diversity.
 Nat Commun. 2015 Jan 30;6:6120.
Nat Commun. 2015 Jan 30;6:6120.
2014年
- Hirokawa M., Morita H., Tajima T., Takahashi A., Ashikawa K., Miya F., Shigemizu D., Ozaki K., Sakata Y., Nakatani D., Suna S., Imai Y., Tanaka T., Tsunoda T., Matsuda K., Kadowaki T., Nakamura Y., Nagai Y., Komuro I., and Kubo M. A genome-wide association study identifies PLCL2 and AP3D1-DOT1L-SF3A2 as new susceptibility loci for myocardial infarction in Japanese.
 Eur J Hum Genet. 2014 Mar;23(3):374-380.
Eur J Hum Genet. 2014 Mar;23(3):374-380.
- Aiba T., Ishibashi K., Wada M., Nakajima I., Miyamoto K., Okamura H., Noda T., Shigemizu D., Satake W., Toda T., Kusano K., Kamakura S., Yasuda S., Sekine A., Miyamoto Y., Tanaka T., Ogawa S., and Shimizu W. Clinical Significance of Whole Exome Analysis Using Next Generation Sequencing in the Genotype-negative Long-QT Syndrome.
 Circulation. 2014 130(Suppl 2) A13817.
Circulation. 2014 130(Suppl 2) A13817.
- Makita N., Yagihara N., Crotti L., Johnson CN., Beckmann BM., Roh MS., Shigemizu D., Lichtner P., Ishikawa T., Aiba T., Homfray T., Behr ER., Klug D., Denjoy I., Mastantuono E., Theisen D., Tsunoda T., Satake W., Toda T., Nakagawa H., Tsuji Y., Tsuchiya T., Yamamoto H., Miyamoto Y., Endo N., Kimura A., Ozaki K., Motomura H., Suda K., Tanaka T., Schwartz PJ., Meitinger T., Kääb S., Guicheney P., Shimizu W., Bhuiyan ZA., Watanabe H., Chazin WJ., and George AL. Novel Calmodulin (CALM2) Mutations Associated with Congenital Arrhythmia Susceptibility.
 Circ Cardiovasc Genet. 2014 Aug;7(4):466-474.
Circ Cardiovasc Genet. 2014 Aug;7(4):466-474.
- Shigemizu D., Abe T., Morizono T., Johnson TA., Boroevich KA., Hirakawa Y., Ninomiya T., Kiyohara Y., Kubo M., Nakamura Y., Maeda S., and Tsunoda T. The Construction of Risk Prediction Models Using GWAS Data and Its Application to a Type 2 Diabetes Prospective Cohort.
 PLoS One. 2014 9(3), e92549.
PLoS One. 2014 9(3), e92549.
- 重水 大智: 遺伝性疾患の網羅的エキソームシーケンスデータの解析法 (2014). 新潟医学会雑誌, 第128巻10号, 496-498.
2013年
- Makita N., Yagihara N., Crotti L., Johnson CN., Beckermann,BM., Shigemizu D., Watanabe H., Ishikawa T., Aiba T., Mastantuono E., Tsunoda T., Nakagawa H., Tsuji Y., Tsuchiya T., Yamamoto H., Miyamoto Y., Endo N., Kimura A., Ozaki K., Motomura H., Suda K., Tanaka T., Schwartz PJ., Meitinge T., Kaeaeb S., Shimizu W., Chazin W., and George AL. CALM2 Mutations Associated With Atypical Juvenile Long QT Syndrome.
 Circulation. 2013 128(22) A13371.
Circulation. 2013 128(22) A13371.
- Imamura M., Shigemizu D., Tsunoda T., Iwata M., Maegawa H., Watada H., Hirose H., Tanaka Y., Tobe K., Kaku K., Kashiwagi A., Kawamori R., and Maeda S. Assessing the Clinical Utility of a Genetic Risk Score Constructed Using 49 Susceptibility Alleles for Type 2 Diabetes in a Japanese Population.
 J Clin Endocrinol Metab. 2013 98(10), E1667-E1673.
J Clin Endocrinol Metab. 2013 98(10), E1667-E1673.
- Shigemizu D., Fujimoto A., Akiyama S., Abe T., Nakano K., Boroevich KA, Yamamoto Y., Furuta M., Kubo M., Nakagawa H., and Tsunoda T. A practical method to detect SNVs and indels from whole genome and exome sequencing data.
 Sci Rep. 2013 3:2161. (natureasia.com注目の論文
Sci Rep. 2013 3:2161. (natureasia.com注目の論文 )
)
2012年
- Nguyen HH., Takata R., Akamatsu S., Shigemizu D., Tsunoda T., Furihata M., Takahashi A., Kubo M., Kamatani N., Ogawa O., Fujioka T., Nakamura,Y., and Nakagawa H. IRX4 at 5p15 suppresses prostate cancer growth through the interaction with vitamin D receptor, conferring prostate cancer susceptibility.
 Hum Mol Genet. 2012 21(9), 2076-2085.
Hum Mol Genet. 2012 21(9), 2076-2085.
- Yoshihara K., Tsunoda T., Shigemizu D., Fujiwara H., Hatae M., Fujiwara H., Masuzaki H., Katabuchi H., Kawakami Y., Okamoto A., Nogawa T., Matsumura N., Udagawa Y., Saito T., Itamochi H., Takano M., Miyagi E., Sudo T., Ushijima K., Iwase H., Seki H., Terao Y., Enomoto T., Mikami M., Akazawa K., Tsuda H., Moriya,T., Tajima T., Inoue I., and Tanaka K. High-risk ovarian cancer based on 126-gene expression signature is uniquely characterized by downregulation of antigen presentation pathway.
 Clin Cancer Res. 2012 18(5), 1374-1385.
Clin Cancer Res. 2012 18(5), 1374-1385.
- Shigemizu D., Hu Z., Hung JH., Huang CL., Wang Y., and DeLisi C. Using functional signatures to identify repositioned drugs for breast, myelogenous leukemia and prostate cancer.
 PLoS Comput Biol. 2012 8(2), e1002347.
PLoS Comput Biol. 2012 8(2), e1002347.
2011年以前
- Takarabe M., Shigemizu D., Kotera M., Goto S., and Kanehisa M. Network-based analysis and characterization of adverse drug–drug interactions.
 J Chem Inf Model. 2011 51(11), 2977-2985.
J Chem Inf Model. 2011 51(11), 2977-2985.
- Moriya Y., Shigemizu D., Hattori M., Tokimatsu T., Kotera M., Goto G., and Kanehisa M. PathPred: an enzyme-catalyzed metabolic pathway prediction server.
 Nucleic Acids Res. 2010 38(suppl 2), W138-W143.
Nucleic Acids Res. 2010 38(suppl 2), W138-W143.
- Takarabe M., Shigemizu D., Kotera M., Goto S., and Kanehisa M. Characterization and classification of adverse drug interactions.
 Genome Inform.2010 22, 167-175.
Genome Inform.2010 22, 167-175.
- Shigemizu D., Araki M., Okuda S., Goto S., and Kanehisa M. Extraction and analysis of chemical modification patterns in drug development.
 J Chem Inf Model. 2009 49(4), 1122-1129.
J Chem Inf Model. 2009 49(4), 1122-1129.
- Shigemizu D., and Maruyama O. Searching for regulatory elements of alternative splicing events using phylogenetic footprinting.
 Algorithms in Bioinformatics Lecture Notes in Computer Science. 2004 Vol. 3240,147-158.
Algorithms in Bioinformatics Lecture Notes in Computer Science. 2004 Vol. 3240,147-158.
招待講演
2025年
- 重水 大智:「 日本人の大規模ヒトゲノム・オミクス解析における認知症研究 」熊本大学大学院生命科学研究部附属健康長寿代謝制御研究センター・国立長寿医療研究センター 第三回共同シンポジウム, 熊本, Mar 22, 2025.
2024年
- 重水 大智:「 日本人の大規模ヒトゲノム・オミクス解析における認知症研究 」第21回糖鎖科学コンソーシアムシンポジウム, 福島, Nov 20, 2024.
- 重水 大智:「 日本人における大規模認知症ゲノム研究の最前線 」国立長寿医療研究センター 公開シンポジウム&レクチャー2024, 大府, June 29, 2024.
- Shigemizu D.:「 A polygenic risk score contributes to identifying individuals with the potential for cognitive function improvement 」2024 ICAH-NCGG-TMIG Annual Meeting, Taiwan, Apr 11, 2024.
2023年
- Shigemizu D.:「 J-MINT study approaching from genomic research 」NJ-FINGERS Symposium, Obu, Dec 15, 2023.
- Shigemizu D.:「 Genomic research on dementia in the Japanese population 」NCGG ICAH TMIG Joint Symposium 2023 IAGG-AOR Sattelite, Tokyo, Oct 17, 2023.
- Shigemizu D., and Ozaki K.:「 Genomic research on dementia in the Japanese population 」Human Genetics Asia 2023, SY7-4, Tokyo, Oct 12, 2023.
- 重水 大智:「 大規模ヒトゲノム・オミクス解析における認知症研究 」ゲノム医科学とバイオインフォマティクスの接点と集学研究, 三島, Feb 22, 2023.
2022年
- Shigemizu D.:「 RNA-sequencing analysis identification of potential biomarkers for diagnosis of sarcopenia 」Sarcopenia Symposium 2022, Seoul, Korea, Dec 20, 2022.
- 重水 大智:「 大規模ヒトゲノム・オミクス解析における認知症原因・感受性遺伝子の同定と発症予測モデルの開発 」熊本大学大学院生命科学研究部附属健康長寿代謝制御研究センター・国立長寿医療研究センター 第一回共同シンポジウム, 熊本, Dec 10, 2022.
- 重水 大智: 「 認知症における日本人大規模ゲノム研究の最前線 」九州大学セミナー, 福岡, Sep 22, 2022.
- 重水 大智: 「 全ゲノム解析による認知症関連レアバリアント解析 」第11回生命医薬情報学連合大会, 大阪, Sep. 14, 2022. (セッションオーガナイザー)
- 重水 大智: 「 大規模ゲノム・オミクス解析を使った認知症研究の最前線 」第765回新潟医学会例会シンポジウム, WEB, May 19, 2022.
- 重水 大智: 「 認知症における日本人大規模ゲノム研究の最前線 」東レ バイオ要素技術連絡会, 鎌倉, Mar 14, 2022.
2021年
- Shigemizu D.: 「 Genetic research in dementia 」An international online congress, Aging Management, WEB, Dec 17, 2021.
- 重水 大智: 「 認知症における日本人大規模ゲノム解析による新規感受性遺伝子の同定 」第94回日本生化学会大会, WEB, Nov 3, 2021.
- 重水 大智: 「 大規模ゲノム・オミクス解析による認知症研究 」日本プロテオーム学会2021年大会, WEB, July 21, 2021.
2020年
- Shigemizu D.: 「 Risk Prediction Models for Dementia Using Genomic Data 」The Sixth ICAH-NCGG Symposium, Taiwan, Oct 22, 2020.
- 重水 大智: 「 大規模ゲノム・オミクス解析による認知症予測研究」第20回日本抗加齢医学会総会, 東京, Sep 26, 2020 (会長特別プログラム).
- 重水 大智: 「 ヒトゲノム・オミクスデータによる認知症血液バイオマーカーの探索と予測モデルの開発 」第93回日本生化学会大会, WEB, Sep 15, 2020.
- 重水 大智: 「 血液由来疾患バイオマーカーの探索と予測モデルの開発 」第9回生命医薬情報学連合大会, WEB, Sep 3, 2020 (セッションオーガナイザー).
- 重水 大智: 「 大規模ゲノム・オミックス解析を使った認知症研究の最前線 」第61回日本神経学会学術大会, 岡山, Aug 31, 2020.
- 重水 大智: 「 ヒトゲノム・オミクスデータを用いた認知症研究の現在 」ゲノム医科学とバイオインフォマティクスの接点と集学研究, 三島, Feb 14, 2020.
2019年
- 重水 大智: 「 ヒトゲノム・オミクスデータを用いた認知症研究の現在 」第8回生命医薬情報学連合大会, 東京, Sep 10, 2019 (セッションオーガナイザー).
- 重水 大智: 「 ヒトゲノム・オミクスデータを用いた認知症研究の現在 」Aging Forum, 大阪, July 13, 2019.
2018年
- 重水 大智: 「 ヒトゲノム・オミクスデータによる疾患原因変異の同定と疾患発症予測モデル開発 」名古屋市立大学医学部セミナー, 名古屋, Nov. 27, 2018.
- 重水 大智: 「 ヒトゲノム・オミクスデータによる疾患原因変異の同定と疾患発症予測モデル開発 」九州工業大学セミナー, 福岡, Nov. 15, 2018.
- 重水 大智: 「 ゲノムデータを用いた疾患原因変異の同定と疾患発症予測モデル開発の現在 」第7回生命医薬情報学連合大会, 鶴岡, Sep 20, 2018.
- Shigemizu D.: 「AI-Aided Genomic Studies for Disease Prediction」2018 AI Future: Application on Geriatrics Care Symposium, Taipei, July 23, 2018.
2017年
- 重水 大智: 「 次世代シークエンサーを用いた臨床研究の実際 」科研費基盤研究S & 第8回生物統計ネットワークシンポジウム 統計科学が切り拓く個別化医療:方法論・実践のフロンティア, 福岡, Mar. 28, 2017.
2016年
- 重水 大智: 「疾患原因となる遺伝子変異同定のためのエクソーム解析の現在」科研費基盤研究Aシンポジウム (I)複雑な生命現象を読み解くための大規模データ解析とモデリング, 久留米, Nov. 7, 2016.
- 重水 大智: 「疾患原因遺伝子同定のためのエクソーム解析の現在」第2回バイオインフォマティクスアゴラ, 東京, July 15, 2016.
2015年以前
- Shigemizu D.: 「Analysis for disease-causing mutations using whole-exome sequencing data」International Symposium on Bioinformatics and its Application, Tokyo Institute of Technology, Tokyo, Japan, Sep. 30, 2014.
- 重水 大智:「エクソームシークエンスデータを使った疾患関連遺伝子同定の解析法」情報・システム研究機構国立遺伝学研究所研究会 ゲノム医科学とバイオインフォマティクスの接点と集学研究, 静岡, Feb. 28, 2014.
- 重水 大智: 「遺伝性疾患の網羅的エキソームシーケンスデータの解析法」シンポジウム 生命システムの理解に向けたバイオインフォーマティクス, 新潟, Nov. 16, 2013.
-
Shigemizu D.: 「New Uses for Old Drugs: Promising Candidates for Breast, Leukemia and Prostate Cancer」National Center for Biotechnology Information, Bethesda, MD, USA, 2010.
- Shigemizu D.: 「Functional analysis at junction sites of alterative splicing regions」Humboldt University, Berlin, Germany, 2005.
発表(口頭:O・ポスター:P)
2024年
- 山川 明子, 菅沼 睦美, 光森 理紗, 新飯田 俊平, 尾崎 浩一, 重水 大智. 網羅的遺伝子発現解析による早期診断のための段階特異的血液バイオマーカーの同定. 第43回日本認知症学会学術集会, 2024, Nov 22, P322, 郡山. P.
- 重水 大智, 中村 昭範, 加藤 隆司, 二橋 尚志, 櫻井 圭太, 武田 章敬, 新畑 豊, 尾崎 浩一, 新飯田 俊平, BATON/STREAM study group. アミロイドβの蓄積速度に関連する全ゲノム解析に基づく遺伝子変異の探索. 第43回日本認知症学会学術集会, 2024, Nov 21, P003, 郡山. O., P.
- Mitsumori R, Asanomi Y, Miyashita A, Morizono T, Hara N, Shigemizu D, Ikeuchi T, Niida S, and Ozaki K. Identification of multiple new loci associated with late-onset Alzheimer’s disease in Japanese. The American Society of Human Genetics, 2024, Nov 6, 5095W, Denver, CO, USA. P.
- Asanomi Y, Kimura T, Shimoda N, Shigemizu D, Niida S, and Ozaki K. Functional analyses for the late-onset Alzheimer’s disease-risk variants of SHARPIN using CRISPR/Cas9-mediated knock-in cells. The American Society of Human Genetics, 2024, Nov 6, 6032W, Denver, CO, USA. P.
- 菅沼 睦美, 古谷 元樹, 細山 徹, 光森 理紗, 大塚 礼, 竹村 真里枝 , 松井 康素 , 佐竹 昭介, 中野 由紀子, 尾崎 浩一 , 重水 大智: 網羅的遺伝子発現解析によるフレイルに関わる血液バイオマーカーの探索. 第11回日本サルコペニア・フレイル学会大会, 2024, Nov 3, P-16-1, 東京. P.
- 細山 徹,漆畑 拓弥,高石 美菜子,佐藤 亜希子,渡邉 剛,竹村 真里枝,重水 大智,関根 圭輔 ,佐竹 昭介: 網加齢変動性マイオカインのαアミラーゼは疾患バイオマーカーになり得る. 第11回日本サルコペニア・フレイル学会大会, 2024, Nov 3, U-3, 東京. O.
- Yamakawa A, Suganuma M, Mitsumori R, Niida S, Ozaki K, and Shigemizu D. Identification of stage-specific blood biomarkers for early diagnosis of Alzheimer’s disease by transcriptome analysis. 1st Asia & Pacific Bioinformatics Joint Conference (APBJC2024), Oct 24, P-84, Okinawa. P.
- Hosoyama T, Kawai-Takaishi M, Takemura M, Watanabe T, Sekine K, Shigemizu D, Satake S. Muscle-derived α-amylase is age-variable myokine and useful for the frailty biomarker. 10th Asian Conference for Frailty and Sarcopenia (ACFS 2024), Oct 11, O01, Bangkok. O.
- Suganuma M, Furutani M, Hosoyama T, Mitsumori R, Otsuka R, Takemura M, Matsui Y, Nakano Y, Niida S, Ozaki K, Satake S, and Shigemizu D. An integrative approach to detect potential blood-based biomarkers for frailty. 10th Asian Conference for Frailty and Sarcopenia (ACFS 2024), Oct 10, P-53, Bangkok. P.
- 光森 理紗, 浅海 裕也, 宮下 哲典, 森園 隆, 原 範和, 重水 大智, 池内 健, 新飯田 俊平, 尾崎 浩一. Genome-wide association study for late-onset Alzheimer’s disease in a Japanese population. 日本人類遺伝学会第69回大会, 2024, Oct 12, O14-2, 札幌. O.
- 山川 明子, 菅沼 睦美, 光森 理紗, 新飯田 俊平, 尾崎 浩一, 重水 大智. Identification of stage-specific blood biomarkers for early diagnosis of Alzheimer’s disease by transcriptome analysis. 日本人類遺伝学会第69回大会, 2024, Oct 12, O14-1, 札幌. O.
- 浅海 裕也, 木村 哲晃, 下田 修義, 重水 大智, 新飯田 俊平, 尾崎 浩一. Functional analyses of the late-onset Alzheimer’s disease-risk SHARPIN variants using CRISPR/Cas9 knock-in cells. 日本人類遺伝学会第69回大会, 2024, Oct 12, P3-02-4, 札幌. P.
- 木村 哲晃, 藤田 康介, 櫻井 孝, 新飯田 俊平, 尾崎 浩一, 重水 大智. Whole genome sequencing reveals East Asian specific rare variants in CDH23 associated with dementia with Lewy bodies. 日本人類遺伝学会第69回大会, 2024, Oct 11, OE5-5, 札幌. O.
- 福永 航也, 重水 大智, 莚田 泰誠. shortHLAseq: 8座のHLA 遺伝子領域をshort-range PCRで増幅するアンプリコンシークエンシング法の開発. 第32回組織適合性学会, 2024, Sep 26, O-08, 名古屋. O.
- Shigemizu D, Nakamura A, Kato T, Nihashi T, Sakurai K, Takeda A, Arahata Y, Ozaki K, Niida S, BATON/STREAM study group. Exploration Of Genetic Variants Associated With The Rate Of Amyloid-β Accumulation Using Whole-genome Sequencing. Alzheimer's Association International Conference (AAIC) Advancements: Modernizing Diagnosis, 2024, Sep 19, P-23, Tokyo. P.
- Kimura T, Suganuma M, Sawamura K, Asanomi Y, Shimoda N, Ogiso N, Hosoyama T, Niida S, Ozaki K, and Shigemizu D. The loss-of-function variant in MFSD3 could play a crucial role in the pathogenesis of dementia with Lewy bodies. Alzheimer's Association International Conference (AAIC), 2024, July 31, P-86694, Philadelphia. P.
- Shigemizu D, Fujita K, Niida S, Ozaki K, Sakurai T, and Arai H. A polygenic risk score contributes to identifying individuals with the potential for cognitive function improvement. Alzheimer's Association International Conference (AAIC), 2024, July 28, P-86348, Philadelphia. P.
- Yamakawa A, Suganuma M, Mitsumori R, Niida S, Ozaki K, and Shigemizu D. Identification of stage-specific blood biomarkers for early diagnosis of Alzheimer’s disease through RNA sequencing analysis. Alzheimer's Association International Conference (AAIC), 2024, July 28, P-85323, Philadelphia. P.
- 吉浦 和宏, 李 嘉琦, 細山 徹, 重水 大智, 竹村 真里枝, 松井 康素, 堀 紀子, 木下 かほり, 大須賀 洋祐, 佐竹 昭介. ロコモフレイル外来受診者におけるGDF-15と身体的フレイル・認知的フレイルとの関連. 第66回日本老年医学会学術集, 2024, June 13, O6-4, 愛知. O.
- 木村 哲晃, 山川 明子, 菅沼 睦美, 新飯田 俊平, 尾崎 浩一, 重水 大智. 日本人の大規模ゲノム解析を通して免疫の側面から見たアルツハイマー病. 第24回日本抗加齢医学会総会, 2024, Jun 1, BP1-7, 熊本. (O, 優秀演題)
- 藤田 康介, 杉本 大貴, 中村 昭範, 重水 大智, 内田 一彰, 松本 奈々恵, 黒田 佑次郎, 横山 陽子, 櫻井 孝, 荒井 秀典. 認知症リスク低減のための多因子介入:効果的な対象集団の探索. 6NCリトリート2024, 2024, Apr 13, P7-12, 東京. P.
- 光森 理紗, 浅海 裕也, 宮下 哲典, 森園 隆, 原 範和, 重水 大智, 池内 健, 新飯田 俊平, 尾崎 浩一. 日本人における孤発性アルツハイマー病の大規模ゲノムワイド関連解析. 6NCリトリート2024, 2024, Apr 13, P11-7, 東京. P.(理事長賞)
2023年
- 木村 哲晃、菅沼 睦美、澤村 嘉代子、浅海 裕也、細山 徹、下田 修義、小木曽 昇、新飯田 俊平、尾崎 浩一、重水 大智. 日本人集団で見つかったレビー小体型認知症に関連する MFSD3 多型の機能解析. 第42回日本認知症学会学術集会, 2023, Nov 25, P346, 奈良. P.
- 山川 明子、光森 理紗、菅沼 睦美、秋山 真太郎、新飯田 俊平、尾崎 浩一、重水 大智. 遺伝子発現データに基づくアルツハイマー病移行予測診断システムの開発. 第42回日本認知症学会学術集会, 2023, Nov 25, P339, 奈良. P.
- Suganuma M, Furutani M, Hosoyama T, Akiyama S, Mitsumori R, Otsuka R, Takemura M, Matsui Y, Nakano Y, Niida S, Ozaki K, Satake S, and Shigemizu D. An integrative approach to detect potential blood-based biomarkers for frailty. The American Society of Human Genetics, 2023, Nov 4, PB3321, WASHINGTON, DC. P.
- Mitsumori R, Asanomi Y, Shigemizu D, Akiyama S, Morizono T, Niida S, and Ozaki K. Identification of an East Asian-specific variant associated with Lewy bodies dementia by genome-wide association study in Japanese subjects. The American Society of Human Genetics, 2023, Nov 3, PB1583, WASHINGTON, DC. P.
- Kimura T, Suganuma M, Hosoyama T, Sawamura K, Shimoda N, Ogiso N, Niida S, Ozaki K, and Shigemizu D. Functional analysis of MFSD3 associated with dementia with Lewy bodies. The American Society of Human Genetics, 2023, Nov 3, PB1109, WASHINGTON, DC. P.
- Asanomi Y, Kimura T, Shimoda N, Shigemizu D, Niida S, and Ozaki K. CRISPR/Cas9-mediated knock-in of late-onset Alzheimer’s disease-risk variant, SHARPIN G186R, lessens the NF-κB pathway and accelerates Aβ secretion. The American Society of Human Genetics, 2023, Nov 2, PB1063, WASHINGTON, DC. P.
- Yamakawa A., Mitsumori R., Suganuma M., Akiyama S., Niida S., Ozaki K., and Shigemizu D.: RNA-seq data analysis identifies blood-based biomarkers for diagnosis and disease progression of Alzheimer's disease. The American Society of Human Genetics, 2023, Nov 2, PB3463, WASHINGTON, DC. P.
- Shigemizu D., Sakai Y., Honjo K., Wakao N., Matsui H., Shimada H., Mitsumori R., Ozaki K., and Watanabe K.: Genome-wide association study for non-specific chronic pain in Japanese elderly. Human Genetics Asia 2023, 2023, Oct 12, P1-10-7, Tokyo. P.
- Suganuma M, Furutani M, Hosoyama T, Akiyama S, Mitsumori R, Otsuka R, Takemura M, Matsui Y, Nakano Y, Niida S, Ozaki K, Satake S, and Shigemizu D. Identification of potential blood-based biomarkers for frailty by using an integrative approach. Human Genetics Asia 2023, 2023, P1-10-6, Oct 12, Tokyo. P.
- Kimura T, Suganuma M, Hosoyama T, Sawamura K, Shimoda N, Ogiso N, Niida S, Ozaki K, and Shigemizu D. Functional analysis of MFSD3 associated with dementia with Lewy bodies. Human Genetics Asia 2023, 2023, Oct 12, P1-10-5, Tokyo. P.
- Mitsumori R, Asanomi Y, Shigemizu D, Akiyama S, Morizono T, Niida S, and Ozaki K. A genome wide association study identifies an East Asian-specific risk variant for Lewy bodies dementia in Japanese. Human Genetics Asia 2023, 2023, Oct 12, P1-10-4, Tokyo. P.
- Asanomi Y, Kimura T, Shimoda N, Shigemizu D, Niida S, and Ozaki K. Knock-in of late-onset Alzheimer’s disease-risk variant SHARPIN G186R lessens NF-κB pathway and accelerates Aβ secretion. Human Genetics Asia 2023, 2023, Oct 12, P1-10-1, Tokyo. P.
- Yamakawa A., Mitsumori R., Suganuma M., Akiyama S., Niida S., Ozaki K., and Shigemizu D.: Exploration of blood-based biomarkers to predict the progression of Alzheimer's disease by RNA-sequencing data analysis. Human Genetics Asia 2023, 2023, Oct 12, P1-05-4, Tokyo. P.
- 木村 哲晃、菅沼 睦美、澤村 嘉代子、細山 徹、下田 修義、小木曽 昇、新飯田 俊平、尾崎 浩一、重水 大智: 日本人集団で見つかったレビー小体型認知症に関連するMFSD3多型の機能解析. 第12回日本認知症予防学会学術集会, 2023, Sep 15, O4-6, 新潟. O.
- 山川 明子、光森 理紗、菅沼 睦美、秋山 真太郎、新飯田 俊平、尾崎 浩一、重水 大智: RNA-seq データ解析に基づく軽度認知機能障害からアルツハイマー型認知症への移行に関与する血液バイオマーカーの探索. 第12回日本認知症予防学会学術集会, 2023, Sep 16, O7-3, 新潟. O.
- 山川 明子、光森 理紗、菅沼 睦美、秋山 真太郎、新飯田 俊平、尾崎 浩一、重水 大智: Identification of blood-based biomarkers associated with conversion from mild cognitive impairment to Alzheimer’s disease by RNA-sequencing data analysis. IIBMP2023, 2023, Sep 7-8, P-106, 柏. P.
- Li J., Yasuoka M., Kinoshita K., Hirano Y., Maeda K., Takemura M., Matsui Y., Hosoyama T., Shigemizu D., Arai H., and Satake S.: Association between spatio-temporal gait parameters and the risk of falls in older adults. IAGG Asia/Oceania Regional Congress 2023, 2023, June 14, P670, Yokohama. P.
- Li J., Hosoyama T., Shigemizu D., Yasuoka M., Kinoshita K., Maeda K., Takemura M., Matsui Y., Arai H., Satake S.: Circulating CXCL9 levels, but not CXCL10 levels, were associated with frailty in older adults. IAGG Asia/Oceania Regional Congress 2023, 2023, June 14, P260, Yokohama. P.
- Ishii K., Ma L., Shigemizu D., Asanomi Y., Nakamura H., Ozaki K., and Watanabe K.: Ferroptotic aspects of cartilage degeneration in mouse osteoarthritis. IAGG Asia/Oceania Regional Congress 2023, 2023, June 13, O1042, Yokohama. O.
- Kimura T., Suganuma M., Sawamura K., Hosoyama T., Ogiso N., Niida S., Ozaki K., and Shigemizu D.: MFSD3 loss of function with dementia with Lewy bodies causes an increase of butyrylcholinesterase activity in the brain. IAGG Asia/Oceania Regional Congress 2023, 2023, June 13, P633, Yokohama. P.
- Mitsumori R., Asanomi Y., Shigemizu D., Akiyama S., Morizono T., Niida S., and Ozaki K.: Genome wide association study identifies new genetic risk loci for dementia with Lewy body in Japanese. IAGG Asia/Oceania Regional Congress 2023, 2023, June 13, P626, Yokohama. P.
- Asanomi Y., Shigemizu D., Akiyama S., Mitsumori R., Niida S., Ozaki K.: East Asian-specific late-onset Alzheimer's disease risk variant alters the endogenous SHARPIN function. IAGG Asia/Oceania Regional Congress 2023, 2023, June 13, P420, Yokohama. P.
- Yamakawa A., Mitsumori R., Akiyama S., Niida S., Ozaki K., and Shigemizu D.: Identification of blood-based biomarkers associated with conversion from mild cognitive impairment to Alzheimer’s disease by RNA-sequencing data analysis. IAGG Asia/Oceania Regional Congress 2023, 2023, June 12, P860, Yokohama. P.
- Suganuma M., Furutani M., Hosoyama T., Akiyama S., Mitsumori R., Otsuka R., Takemura M., Matsui Y., Nakano Y., Niida S., Ozaki K., Satake S., and Shigemizu D.: Identification of blood-based biomarkers for early diagnosis of frailty through a combined analysis of the clinical data, gene-expression data, and aging-related factors. IAGG Asia/Oceania Regional Congress 2023, 2023, June 12, P632, Yokohama. P.
- 光森 理紗, 浅海 裕也, 重水 大智, 秋山 真太郎, 森園 隆, 新飯田 俊平, 尾崎 浩一: 日本人および民族間横断的ゲノムワイド関連解析によるレビー小体認知症感受性座位の探索. 第31回日本医学会総会2023東京, 2023, Apr 22, P11-4, 東京. P.(理事長賞)
2022年
- 古谷 元樹, 森園 隆, 光森 理紗, 呉谷 文, 中野 由紀子, 新飯田 俊平, 重水 大智, 尾崎 浩一: 日本人集団におけるQTL解析. 日本人類遺伝学会第67回大会, 2022, Dec 15, PE2-5, 横浜. P.
- 光森 理紗, 浅海 裕也, 重水 大智, 秋山 真太郎, 森園 隆, 新飯田 俊平, 尾崎 浩一: 日本人および民族間横断的ゲノムワイド関連解析によるレビー小体認知症感受性座位の探索. 日本人類遺伝学会第67回大会, 2022, Dec 15, P-12-12, 横浜. P.
- 浅海 裕也, 重水 大智, 秋山 真太郎, 光森 理紗, 新飯田 俊平, 尾崎 浩一: 日本人における遅発性アルツハイマー病関連遺伝子SHARPINの同定と解析. 日本人類遺伝学会第67回大会, 2022, Dec 15, P-12-10, 横浜. P.
- 木村 哲晃, 浅海 裕也, 秋山 真太郎, 光森 理紗, 森園 隆, 新飯田 俊平, 尾崎 浩一, 重水 大智: 全ゲノムシーケンスによるアルツハイマー病に関連したMLKL遺伝子の日本人に特異的な新規希少バリアントの発見. 日本人類遺伝学会第67回大会, 2022, Dec 16, P-21-1, 横浜. P.
- 呉谷 文, 森園 隆, 秋山 真太郎, 新飯田 俊平, 尾崎 浩一, 重水 大智: 日本人大規模データを用いたアルツハイマー病トランスクリプトームワイド関連解析. 第45回日本分子生物学会年会, 2022, Nov 30, 1P-718, 幕張. P.
- 古谷 元樹, 菅沼 睦美, 秋山 真太郎, 光森 理紗, 細山 徹, 竹村 真里枝, 松井 康素, 佐竹 昭介, 中野 由紀子, 尾崎 浩一, 重水 大智: 網羅的遺伝子発現解析によるサルコペニアに関わるバイオマーカーの探索. 第9回日本サルコペニア・フレイル学会大会, 2022, Oct. 29-30, I-064, 草津, P.
- Furunani M., Suganuma M, Akiyama S., Mitsumori R., Hosoyama T., Takemura M., Matsui Y., Satake S., Nakano Y., Niida S., Ozaki K., and Shigemizu D.: Identification of potential blood biomarkers for Sarcopenia through RNA sequencing analysis. The 8th Asian Conference for Frailty and Sarcopenia, 2022, Oct. 28, P07-2, Nagoya, P.
- Asanomi Y., Shigemizu D., Akiyama S., Mitsumori R., Niida S., and Ozaki K.: Identification of a novel functional missense variant associated with late-onset Alzheimer's disease in Japanese. 第11回生命医薬情報学連合大会, 2022, Sep. 14, P-82, Osaka, P.
2021年
- 馬 凌云, 重水 大智, 中村 博幸, 尾崎 浩一, 渡邉 研: 変形性関節症自然発症マウスモデルSTR/ortの軟骨変性に関わる遺伝子変異の同定とferroptosisの関与. 第94回日本生化学会大会, 2021, Nov 3, P-503, WEB, P.
- 浅海 裕也, 重水 大智, 秋山 真太郎, 宮下 哲典, 光森 理紗, 原 範和, 池内 健, 新飯田 俊平, 尾崎 浩一: 遅発性アルツハイマー病関連遺伝子SHARPINの疾患リスクとなる新規機能的ミスセンスバリアントの同定. The Japan Society of Human Genetics, 2021, Oct. 15, O8-5, 横浜, O.
- 光森 理紗, 浅海 裕也, 重水 大智, 秋山 真太郎, 森園 隆, 寺尾 知可史, 新飯田 俊平, 尾崎 浩一: 日本人および民族横断的ゲノムワイド関連解析による認知症の感受性座位の探索. The Japan Society of Human Genetics, 2021, Oct. 15, P27-1, 横浜, P.
- 古谷 元樹, 岡村 祥央, 森園 隆, 重水 大智, 新飯田 俊平, 中野 由紀子, 尾崎 浩一: 日本人集団における頻脈誘発性心筋症のゲノムワイド関連解析. The Japan Society of Human Genetics, 2021, Oct. 14, P16-4, 横浜, P.
2020年
- 光森 理紗, 浅海 裕也, 重水 大智, 秋山 真太郎, 森園 隆, 新飯田 俊平, 尾崎 浩一: ゲノムワイド関連解析による4種類の認知症における感受性座位の探索. The Japan Society of Human Genetics, 2020, P8-1, WEB, P.
- Asanomi Y., Shigemizu D., Mitsumori R., Niida S., and Ozaki K.: Identification of the novel rare variants in SHARPIN associated with the risk of late-onset Alzheimer's disease. The Japan Society of Human Genetics, 2020, OE11-5, WEB, O.
- Kikuchi M., Miyashita A., Hara N., Shigemizu D., Ozaki K., Niida S., Ikeuchi T., Nakaya A.: Polygenic analysis of late-onset Alzheimer’s disease in a Japanese population. 2020 Alzheimer's Association International Conference, 2020, July 27, Virtual Event, P.
- Ma L., Shigemizu D., Akiyama S., Jokaji R., Nakamura H., Ozaki K., Watanabe K.: Identification Of A Gene Mutation Involved In Cartilage Degeneration In Str/ort, A Mouse Line Developing Spontaneous Osteoarthritis. ORS 2020 Annual Meeting, 2020, Feb. 11, PS1-002-581, Phoenix, Arizona, USA, P.
2019年
- 馬 凌云, 重水 大智, 秋山 真太郎, 尾崎 浩一, 渡辺 研: 変形性関節症自然発症マウスのゲノム解析. The Japan Society of Human Genetics, 2019, Nov. 8, P34-2, Nagasaki, Japan, P.
- Wong JH., Shigemizu D., Yoshii Y., Akiyama S., Tanaka A., Nakagawa H., Narumiya S., and Fujimoto A.: Identification of intermediate-sized deletions and inference of their impact on gene expression in a human population. The Japan Society of Human Genetics, 2019, Nov. 8, APCHG-4, Nagasaki, Japan, O.
- Shigemizu D., Akiyama S., Higaki S., Sugimoto T., Sakurai T., Niida S., and Ozaki K.: Prognosis prediction model for Alzheimer’s disease conversion from mild cognitive impairment using integrative multi-omics data. The Japan Society of Human Genetics, 2019, Nov. 9, O27-4, Nagasaki, Japan, O.
- Asanomi Y., Shigemizu D., Miyashita A., Mitsumori R., Mori T., Hara N., Ito K., Niida S., Ikeuchi T., and Ozaki K.: A nonsynonymous variant of SHARPIN attenuates the inflammatory response and associates with late-onset Alzheimer's disease. The Japan Society of Human Genetics, 2019, Nov. 7, BP-1, Nagasaki, Japan, P.
- Mori T., Shigemizu D., Asanomi Y., Mitsumori R., Niida S., and Ozaki K.: Functional analysis for the variants associated with the risk of late-onset Alzheimer’s disease. The Japan Society of Human Genetics, 2019, Nov. 7, P1-2, Nagasaki, Japan, P.
- Mitsumori R., Shigemizu D., Akiyama S., Asanomi Y., Mori T., Niida S., and Ozaki K.: Genome wide association study for dementias in a Japanese population. The Japan Society of Human Genetics, 2019, Nov. 7, O8-4, Nagasaki, Japan, O.
- 馬 凌云, 重水 大智, 秋山 真太郎, 定梶 嶺, 中村 博幸, 尾崎 浩一, 渡辺 研: 変形性関節症を自然発症するSTR/ortマウスのゲノム解析による軟骨変性関連遺伝子の同定. The 42 Annual Meeting of the Molecular Biology Society of Japan, 2019, Dec. 4, 2P-0270, Fukuoka, P.
- 岩波 礼将, 横井 勇人, 秋山 真太郎, 重水 大智, 荒木 夏生, 二宮 尚, 松田 勝, 鈴木 徹, 下田 修義: Role of gene body methylation revealed by a viable zebrafish DNA methyltransferase mutant. The 42 Annual Meeting of the Molecular Biology Society of Japan, 2019, Dec. 4, 2P-0102, Fukuoka, P.
- Mitsumori R., Shigemizu D., Akiyama S., Asanomi Y., Mori T., Niida S., and Ozaki K.: 日本人における5種認知症のゲノムワイドな関連解析. The 42 Annual Meeting of the Molecular Biology Society of Japan, 2019, Dec. 3, 1P-0627, Fukuoka, P.
- Akiyama S., Higaki S., Niida S., Ozaki K., and Shigemizu D.: JAMIR-eQTL: Japanese genome-wide identification of microRNA expression quantitative trait loci. The 42 Annual Meeting of the Molecular Biology Society of Japan, 2019, Dec. 5, 3P-0651, Fukuoka, P.
- Asanomi Y., Shigemizu D., Sakurai T., Ozaki K., Ochiya T., and Niida S.: Multiclass classification model for dementia-type prediction using serum microRNA biomarkers. Informatics In Biology, Medicine and Pharmacology (IIBMP2019), 2019, Sep. 10, P-70, Tokyo, P.
- Akiyama S., Higaki S., Niida S., Ozaki K., and Shigemizu D.: JAMIR-eQTL: Japanese genome-wide identification of microRNA expression quantitative trait loci. Informatics In Biology, Medicine and Pharmacology (IIBMP2019), 2019, Sep. 10, P-28, Tokyo, P.
- Mitsumori R., Shigemizu D., Akiyama S., Asanomi Y., Mori T., Niida S., and Ozaki K.: Genome wide association study for dementias in a Japanese population. The American Society of Human Genetics, 2019, Oct. 19, P2082W, HOUSTON, TX, USA, P.
- Asanomi Y., Shigemizu D., Miyashita A., Mitsumori R., Mori T., Hara N., Ito K., Niida S., Ikeuchi T., and Ozaki K.: Whole exome sequencing identifies a rare functional variant SHARPIN G186R associated with increased risk of late-onset Alzheimer's disease. The American Society of Human Genetics, 2019, Oct. 19, P2067W, HOUSTON, TX, USA, P.
- Shigemizu D., Akiyama S., Asanomi Y., Boroevich KA., Sharma A., Tsunoda T., Sakurai T., Ozaki K., and Niida S.: A comparison of machine learning classifiers for dementia with Lewy bodies using miRNA expression data. The American Society of Human Genetics, 2019, Oct. 17, 1471T, HOUSTON, TX, USA, P.
- 岩波 礼将, 横井 勇人, 秋山 真太郎, 重水 大智, 荒木 夏生, 二宮 尚, 松田 勝, 鈴木 徹, 下田 修義: ジーンボディにおけるDNAメチル化の機能的役割. 第13回日本エピジェネティクス研究会年会, 2019, May 28-29, Yokohama, Japan, P.
- Shigemizu D., Akiyama S., Asanomi Y., Boroevich KA., Sharma A., Tsunoda T., Matsukuma,K., Ichikawa M., Sudo H., Takizawa S., Sakurai T., Ozaki K., and Niida S.: The construction of risk prediction models for dementia with supervised principal component analysis using miRNA expression data. 第6回JMACシンポジウム, 2019, Jan. 24-25, Tokyo, Japan, P.
- Asanomi Y., Shigemizu D., Kayano M., Matsukuma K., Ichikawa M., Sudo H., Takizawa S., Sakurai T., Ozaki K., and Niida S.: Exploration of serum microRNA biomarkers for dementia-risk prediction. 第6回JMACシンポジウム, 2019, Jan. 24-25, Tokyo, Japan, P.
2018年
- 芦崎 晃一, 川崎 洋, 川上 英良, 重水 大智, 桜田 一洋, 角田 達彦, 海老原 全, 天谷 雅行: 医療データ駆動型研究を推進する医療データ収集システムの構築と導入効果、第38回日本医療情報学連合大会, 2018, Nov. 23, 2-I-3-2, Fukuoka, Japan, O.
- Reddy HM., Sharma A., Dehzangi A., Shigemizu D., Chandra AA. and Tsunoda T.: GlyStruct: Glycation prediction using structural properties of amino acid residues. International Conference on Bioinformatics (INCOB), 2018, Sep. 26, SFIB-5, New Delhi, INDIA, O.
- Shigemizu D., Akiyama S., Asanomi Y., Boroevich KA., Sharma A., Tsunoda T., Matsukuma,K., Ichikawa M., Sudo H., Takizawa S., Sakurai T., Ozaki K., and Niida S.: The construction of risk prediction models for dementia with supervised principal component analysis using miRNA expression data. The American Society of Human Genetics, 2018, Oct. 19, P2543F, San Diego, CA, USA, P.
- Mori T., Shigemizu D., Akiyama S., Mitsumori R., Asanomi Y., Niida S., and Ozaki K.: Expression quantitative trait loci analysis with blood cells from late-onset Alzheimer's disease patients in Japanese. The Japan Society of Human Genetics, 2018, Oct. 12, P-260, Yokohama, Japan, P.
- Mitsumori R., Asanomi Y., Shigemizu D., Mori T., Akiyama S., Niida S., and Ozaki K.: Genome wide association study for four types of dementia in Japanese population. The Japan Society of Human Genetics, 2018, Oct. 12, P-167, Yokohama, Japan, P.
- Wong JH., Akiyama S., Nakagawa H., Shigemizu D., and Fujimoto A.: Identification and characterization of intermediate-sized deletions within a Japanese population. The Japan Society of Human Genetics, 2018, Oct. 11, P-120, Yokohama, Japan, P.
- Miya F., Shigemizu D., Kanemura Y., Saitoh S., Okamoto N., Kato M., Matsunaga T., Mutai H., Kosaki K., and Tsunoda T.: Beyond the limitations of exome analysis for genetic disorders. The Japan Society of Human Genetics, 2018, Oct. 11, P-118, Yokohama, Japan, P.
- Asanomi Y., Shigemizu D., Mitsumori R., Mori T., Niida S., and Ozaki K.: Association study of novel risk rare variants of late-onset Alzheimer's disease. The Japan Society of Human Genetics, 2018, Oct. 12, BO-04, Yokohama, Japan, O.
- Shigemizu D., Akiyama S., Asanomi Y., Boroevich KA., Sharma A., Tsunoda T., Matsukuma,K., Ichikawa M., Sudo H., Takizawa S., Sakurai T., Ozaki K., and Niida S.: The construction of risk prediction models for dementia with supervised principal component analysis using miRNA expression data. The Japan Society of Human Genetics, 2018, Oct. 12, O-102, Yokohama, Japan, O.
- Asanomi Y., Shigemizu D., Kayano M., Matsukuma K., Ichikawa M., Sudo H., Takizawa S., Ozaki K., and Niida S.: Exploration of serum microRNA biomarkers for dementia risk prediction. 第10回日本RNAi研究会 / 第5回日本細胞外小胞学会JSEV, 2018, Aug 30, P-64, Hiroshima, Japan, P.
- Shigemizu D., Akiyama S., Asanomi Y., Boroevich KA., Sharma A., Tsunoda T., Matsukuma K., Ichikawa M., Sudo H., Takizawa S., Sakurai T., Ozaki K., and Niida S.: The construction of risk prediction models for dementia with supervised principal component analysis using miRNA expression data. 第10回日本RNAi研究会 / 第5回日本細胞外小胞学会JSEV, Hiroshima, 2018, Aug 30, P-60, P.
- Shigemizu D.: Clinical study using Next generation sequencing. Japanese Society of Bioinformatics, Kagoshima, 2018, May 25, O.
- Okamoto K., Shigemizu D., Okano T., Yeh TW., Takashima T., Yamashita M., Tanita K., Ono S., Mitsuiki N., Naruto T., Okada S., Takagi M., Mori M., Kanegane H., Tsunoda T., Imai K., and Morio T.: Whole exome sequence analysis using the known and candidate genes for primary immunodeficiency diseases. The 2nd Scientific Congress of Asia-Pacific Society for Immunodeficiencie, 2018, May 7, Chongqing, China, P.
- Yagihara N., Watanabe Y., Makita N., Horie M., Simizu W., Ono S., Hasegawa K., Aiba T., Tanaka T., Tsunoda T., Shigemizu D., and Minamino T.:Identification of Mutations in Causative Genes for Cardiomyopathies in Patients with Arrhythmia Syndromes and Structurally Normal Heart. The 82th Annual Scientific Meeting of the Japanese Circulation Society, 2018, Mar. 24, Osaka, Japan, P.
2017年
- Watanabe R., Ohno S., Aiba T., Ishikawa Nakano Y., Aizawa Y., Hayashi K., Murakoshi N., Nakajima T., Yagihara N., Shigemizu D., Mishima H., Sudo T., Higuchi C., Takahashi A., Sekine A., Minamino N., Makiyama T., Tanaka Y., Murata H., Hayashi M., Iwasaki Y., Watanabe A., Tachibana M., Morita H., Miyamoto Y., Yoshiura K., Tsunoda T., Watanabe H., Kurabayashi M., Nogami A., Kihara Y., Makita N., Shimizu W., Horie M., and Tanaka T.: Targeted deep sequencing reveals novel mutations in Japanese long QT syndrome patients. The Japan Society of Human Genetics, 2017, P-162, Kobe, Hyogo, Japan, P.
- Miya F., Shigemizu D., Kanemura Y., Saitoh S., Okamoto N., Kato M., Yamasaki M., Matsunaga T., Mutai H., and Kosaki K.: Development and assessment of a pathogenic mutations search method for subjects where exome has previously failed. The Japan Society of Human Genetics, 2017, O-98, Kobe, Hyogo, Japan, O.
- Shigemizu D., Miya F., Akiyama S., Okuda S., Boroevich KA., Fujimoto A., Nakagawa H., Ozaki K., Niida S., Kanemura Y., Okamoto N., Saitoh S., Kato M., Yamasaki M., Matsunaga T., Mutai H., Kosaki K., and Tsunoda T.: An accurate intermediate-size indel detection tool for discovery of mutations associated with human disease. The Japan Society of Human Genetics, 2017, O-97, Kobe, Hyogo, Japan, O.
- Asanomi Y., Shigemizu D., Nagata Y., Mitsumori R., Mori T., Niida S., and Ozaki K.: Exome sequencing analyses for late-onset Alzheimer's disease in Japanese. The Japan Society of Human Genetics, 2017, O-91, Kobe, Hyogo, Japan, O.
- Mitsumori R., Asanomi Y., Shigemizu D., Nagata Y., Mori T., Akiyama S., Niida S., and Ozaki K.: Genome wide association study for late-onset Alzheimer's Disease in a Japanese population. The Japan Society of Human Genetics, 2017, O-90, Kobe, Hyogo, Japan, O.
- Shigemizu D., Miya F., Akiyama S., Okuda S., Boroevich KA., Fujimoto A., Nakagawa H., Ozaki K., Niida S., Kanemura Y., Okamoto N., Saitoh S., Kato M., Yamasaki M., Matsunaga T., Mutai H., Kosaki K., and Tsunoda T.: Development of an accurate intermediate-size indel detection tool for discovery of mutations associated with human disease. Informatics In Biology, Medicine and Pharmacology (IIBMP2017), 2017, O1-1, Sapporo, O,P, 研究奨励賞.
- Shigemizu D., Miya F., Akiyama S., Okuda S., Boroevich KA., Fujimoto A., Nakagawa H., and Tsunoda, T.: IMSindel: An accurate intermediate-size indel detection tool incorporating de novo assembly and gapped global-local alignment into spit reads. The American Society of Human Genetics, 2017, P1374F, Orland, FL, USA, P.
- Watanabe, R., Ohno S., Aiba T., Ishikawa T., Nakano Y., Aizawa Y., Hayashi K., Murakoshi N., Nakajima T., Yagihara N., Shigemizu D., Mishima H., Sudo T., Higuchi C., Takahashi A., Sekine A., Minamino N., Makiyama T., Tanaka Y., Murata H., Hayashi M., Iwasaki Y., Watanabe A., Tachibana M., Morita H., Miyamoto Y., Yoshiura K., Tsunoda T., Watanabe H., Kurabayashi M., Nogami A., Kihara Y., Makita N., Shimizu W., Horie M., and Tanaka T.: Identification of novel long QT syndrome-associated mutations by targeted sequencing analyses. European Human Genetics Conference, 2017, Copenhagen, Denmark, P.
- 宮 冬樹, 重水 大智, 齋藤 伸治, 須藤 章, 中川 英刀, 奥田 修二郎, 岡本 伸彦, 加藤 光広, 山崎 麻美, Keith A. Boroevich, 金村 米博, 小崎 健次郎, 角田 達彦: 既存のexome解析では疾患原因変異同定が困難な検体の原因変異探索手法の開発. NGS現場の会第5回研究会, 2017, 仙台, P.
2016年
- Matsukura M., Sindo S., Kimura S., Yamamoto K., Hoshina K., Mitsui J., Tsuji S., Shigemizu D., Tsunoda T., Ozaki K., Tanaka T., and Watanabe T.: Investigation of Disease Associated Genetic Factors of Peripheral Arterial Disease in a Japanese Population. The 57th Annual Meeting of Japanese College of Angiology, 2016, Nara, O.
- Shigemizu D., Miya F., Fujimoto A., Boroevich KA., Okuda S., and Tsunoda T: Development of a long indel detection method using the realignment of the misaligned reads. The American Society of Human Genetics, 2016, P1867W, Vancouver, Canada, P.
- Satake W., Suzuki Y., Shigemizu D., Takahashi A., Yamamoto K., Murata M., Hattori N., Tsunoda T., Tsuji S., Kubo M., Sugano S., Matsumoto N., and Toda T.: Exome Association study and 2nd SNP-GWAS of Parkinsion's disease in Japan. The 13th International Congress of Human Genetics, 2016, Kyoto, Japan, O.
- Shigemizu D., Momozawa Y., Abe T., Morizono T., Boroevich KA., Takata S., Ashikawa K., Kubo M., and Tsunoda T.: Performance comparison of four commercial human whole-exome capture platforms. The 13th International Congress of Human Genetics, 2016, P111, Kyoto, Japan, P.
2015年
- Satake W., Suzuki Y., Shigemizu D., Cha PC., Tsuji S., Tsunoda T., Kubo M., Sugano S., Matsumoto N., and Toda T.: Exome Association study and SNP-GWAS of Parkinson’s disease. The 11th International workshop on Advanced Genomics, 2015, Tokyo, Japan, O.
- Shigemizu D., Momozawa Y., Abe T., Morizono T., Boroevich KA., Takata S., Ashikawa K., Kubo M., and Tsunoda T.: Performance comparison of four commercial human whole-exome capture platforms. Informatics in Biology, Medicine and Pharmacology, 2015, P13, Kyoto, Japan, P.
- Shigemizu D., Momozawa Y., Abe T., Morizono T., Boroevich KA., Takata S., Ashikawa K., Kubo M., and Tsunoda T.: Performance comparison of four commercial human whole-exome capture platforms. The American Society of Human Genetics, 2015, 1876W, Baltimore, MD, USA, P.
2014年
- Shigemizu D., Ozaki K., Fujimoto A., Miya F., Akiyama S., Boroevich KA., Abe T., Nakano K., Kochi Y., Satake W., Toda T., Aiba T., Miyamoto Y., Shimizu W., Nakagawa H.,Tsunoda T., and Tanaka T.: Exome sequencing of 35 Japanese families with Long QT syndrome reveals candidate pathogenic mutations in calmodulin-interacting genes. The Japan Society of Human Genetics, 2014, 1BO-3, Funabashi, Tokyo, Japan, 大会賞候補, O.
- Satake W., Shigemizu D., Suzuki Y., Yamamoto K., Tomiyama H., Takahashi A., Murata M., Hattori N., Tanaka T., Tsunoda T., Kubo M., Tsuji S., Matsumoto N., Sugano S. and Toda T.: Exome association study and 2nd SNP-GWAS of Parkinson's disease. The Japan Society of Human Genetics, 2014, 3O8-5, Funabashi, Tokyo, Japan, O.
- Satake W., Shigemizu D., Suzuki Y., Yamamoto K., Tomiyama H., Yamamoto M., Murata M., Hattori N., Tsunoda T., Kubo M., Tsuji S., Nakamura Y., Sugano S. and Toda T.: Exome association study and 2nd SNP-GWAS of Parkinson's disease. The American Society of Human Genetics, 2014, 1232M, San Diego, CA, USA, P.
- Shigemizu D., Fujimoto A., Akiyama S., Abe T., Nakano K., Boroevich KA., Yamamoto Y., Furuta M., Kubo M., Nakagawa H., and Tsunoda T.: A practical method to detect SNVs and indels from whole genome and exome sequencing data and an importance of in-house data for variant filtering. The American Society of Human Genetics, 2014, 1574T, San Diego, CA, USA, P.
- Fujimoto A., Furuta M., Shiraishi Y., Nguyen HH., Shigemizu D., Gotoh K., Kawakami Y., Nakamura T., Ueno M., Arizumi S., Shibata T., Ojima H., Shimada K., Hayami S., Shigekawa Y., Aikata H., Arihiro K., Ohdan H., Marubashi S., Yamada T., Ishikawa O., Kubo M., Hirano S., Yamamoto M., Yamaue H., Chayama K., Miyano S., Tsunoda T., and Nakagawa H.: Analysis of mutational landscape and genetic heterogeneity in liver cancer with whole genome sequencing. The American Society of Human Genetics, 2014, 134, San Diego, CA, USA, O.
2013年
- Shigemizu D., Fujimoto A., Akiyama S., Abe T., Nakano K., Boroevich KA, Kubo M., Nakagawa H., and Tsunoda T.: A practical method to detect SNVs and indels from whole genome and exome sequencing data. Biochemistry and Molecular Biology, 2013, P2-0056, Kobe, Japan, P.
- Shigemizu D., Fujimoto A., Akiyama S., Abe T., Nakano K., Boroevich KA, Kubo M., Nakagawa H., and Tsunoda T.: A practical method to detect SNVs and indels from whole genome and exome sequencing data. The Japan Society of Human Genetics, 2013, O20, Sendai, Miyagi, Japan, O.
2012年以前
- Shigemizu D., Abe T., Morizono T., Johnson TA., Boroevich KA., Kubo M., Nakamura Y., Maeda S., and Tsunoda T.: The effect of genetic and interaction factors for risk prediction in type 2 diabetes. Biochemistry and Molecular Biology, 2012, 1P-0033, Fukuoka, Japan, P, O.
- Shigemizu D., Abe T., Morizono T., Johnson TA., Boroevich KA., Kubo M., Nakamura Y., Maeda S., and Tsunoda T.: The effect of genetic and interaction factors for risk prediction in type 2 diabetes. The American Society of Human Genetics, 2012, San Francisco, CA, USA, P.
- Takarabe M., Shigemizu D., Kotera M., Goto S., and Kanehisa M.,: Network-based Analysis and Characterization of Drug Interactions. The 110th ICR Annual Symposium, 2010, Kyoto, Japan, P.
- Shigemizu D., Araki M., Goto S., and Kanehisa M.: Analysis of chemical modification patterns extracted from KEGG DRUG structure maps. 9th International Workshop on Bioinformatics & Systems Biology, 2009, Boston, USA, O.
- Shigemizu D., Araki M., Okuda S., Goto S., and Kanehisa M.: Extraction of the drug modification patterns from the KEGG DRUG database. 21st COE program - Symposium on Bioinformatics and Chemical Genomics, 2007, P-30, Kyoto, Japan, P.
- Shigemizu D., Araki M., Okuda S., Goto S., and Kanehisa M.: Extraction of the drug design library from the KEGG DRUG database. 21st COE program Working Committee of Open International Symposium, 2007, O-01, Kyoto, Japan, O.
- Shigemizu D., Araki M., Okuda S., Goto S., and Kanehisa M.: Extraction of chemical modification patterns based on KEGG DRUG structure map. Biochemistry and Molecular Biology, 2007, 4P-1203, Yokohama, Japan, P.
- Shigemizu D., Okuda S., Araki M., Goto S., and Kanehisa M.: Construction of the drug substructure library from the KEGG DRUG database. 6th International Workshop on Bioinformatics & Systems Biology, 2006, Boston, USA, P.
- Shigemizu D., Okuda S., Yoshizawa A., Ito M., Hattori M., Goto S., and Kanehisa M.: Association analysis of alternative splicing and the protein function variations. The 28th Annual Meeting of the Molecular Biology Society of Japan, 2005, 2P-0765, Fukuoka, Japan, P.
- Maruyama O., and Shigemizu D.: Searching for Regulatory Elements of Alternative Splicing Using Phylogenetic Footprinting. 13th Annual International Conference on Intelligent Systems for Molecular Biology (ISMB), 2005, D-34, Michigan, USA, P.
- Shigemizu D., and Maruyama, O.: Searching for Regulatory Elements of Alternative Splicing Using Phylogenetic Footprinting. ALGO 2004 Symposium and Workshops, 2004, Bergen, Norway, O.
先頭へ戻る
教育
2019年
- 東京医科歯科大学 医歯学総合研究科 医歯科理工学専攻 修士課程 疾患オミックス情報学特論 (前期)
2018年
- 東京医科歯科大学 医歯学総合研究科 医歯科理工学専攻 修士課程 疾患オミックス情報学特論 (前期)
2017年
- TMDU, Master course, Disease OMICS Informatics (後期)
- 東京医科歯科大学 医歯学総合研究科 医歯科理工学専攻 修士課程 疾患オミックス情報学特論 (前期)
- 東京医科歯科大学 医歯学総合研究科 医歯科理工学専攻 修士課程 バイオインフォマティクス (前期)
2016年
- TMDU, Master course, Bioinformatics (後期)
- 東京医科歯科大学 医歯学総合研究科 生命理工学系専攻 博士課程 生命情報科学特論 (後期)
- 東京医科歯科大学 医歯学総合研究科 医歯科理工学専攻 修士課程 疾患オミックス情報学特論 (前期)
- 東京医科歯科大学 医歯学総合研究科 医歯科理工学専攻 修士課程 バイオインフォマティクス (前期)
2015年
- 東京医科歯科大学 医歯学総合研究科 医歯科理工学専攻 修士課程 疾患オミックス情報学特論 (後期)
- 東京医科歯科大学 医歯学総合研究科 医歯科理工学専攻 修士課程 バイオインフォマティクス (前期)
先頭へ戻る
外部資金
2025年
- 公益財団法人ソルト・サイエンス研究財団 一般公募助成研究:医学分野 2025年-2026年 分担「マグネシウムトランスポーターSLC41A3の骨格筋における役割とサルコペニアとの関連性の解明 」
2024年
- 長寿医療研究開発費 2024年-2027年 代表「大規模ゲノム解析における老年病発症関連遺伝子変異の同定と人工知能による発生リスク予測モデルの開発」
- 公益財団法人 三井住友海上福祉財団 2024年-2025年 代表「全ゲノム解析を基盤としたレビー小体型認知症の病態メカニズムの解明」
- 公益財団法人 中冨健康科学振興財団 2024年-2026年 代表「全ゲノム解析からサルコペニア診断に有効なバイオマーカーの探索と病態メカニズムの 解明」
2023年
- 特別長寿医療研究開発費 2023年-2026年 分担 代表「認知症発症関連遺伝子変異の機能解析と基盤開発」
2022年
2021年
- 国立高度専門医療研究センター 横断的研究推進費研究課題 2021年- 分担「ロコモフレイル外来患者のフレイルバイオマーカーの探索研究」
- 国立高度専門医療研究センター 横断的研究推進費研究課題 2021年- 分担「医療前のライフログデータおよび健診結果を活用する予測先制医療のための研究」
- 文部科学省科学研究費補助金 基盤研究(B) 2021年-2025年 代表「マルチオミクス統合解析によるアルツハイマー病移行予測診断システムの開発」
- 長寿医療研究開発費 2021年-2023年 代表「認知症発症関連因子・機能モジュールの探索と人工知能を用いた発症予測モデルの開発」
- 長寿医療研究開発費 2021年-2023年 分担「サルコペニアバイオマーカーの同定」
2020年
- AMED 認知症研究開発事業 2020年-2023年 分担「血液バイオマーカーを用いた超早期アルツハイマー病コホートの構築」
- 公益財団法人 大幸財団 自然科学系学術研究助成 2020年-2021年 代表「ヒトゲノム・オミクスデータを用いたアルツハイマー病移行診断予測システムの開発」
- 公益財団法人 長寿科学振興財団 長寿科学研究者支援事業 研究継続 2020年 代表「アルツハイマー病発症・移行リスク因子の同定と早期予測診断システムの開発」
2019年
- 長寿医療研究開発費 2019年-2021年 分担「高齢者における非特異的慢性疼痛の病態解明の基盤形成」
- 公益財団法人 長寿科学振興財団 長寿科学研究者支援事業 2019年 代表「アルツハイマー病発症・移行リスク因子の同定と早期予測診断システムの開発」
- AMED 認知症対策官民イノベーション実証基盤整備事業 2019年-2021年 分担「認知症予防を目指した多因子介入によるランダム化比較研究」
- 文部科学省科学研究費補助金 基盤研究(B) 2019年-2021年 分担「小児急性リンパ性白血病発症にかかわる遺伝学的背景の検討」
2018年
- 公益財団法人 武田科学振興財団 医学系研究奨励 2018年-2019年 代表「PIB-PET情報と全ゲノム情報から アルツハイマー病の発症・進行規定因子の解明」
- 理研 理事長裁量経費 2018年 代表「リウマチ患者の末梢血RNASeq解析による薬剤効果予測」
- 長寿医療研究開発費 2018年-2020年 代表「ゲノムデータ等を用いた疾患関連因子の網羅的探索と疾患発症リスク予測モデルの開発 」
- 厚生労働省 認知症政策研究事業 2018年-2020年 分担「日本人認知症ゲノム解析を出発点としたオミックス-臨床情報統合解析による疾患関連パスウェイの解析から診断、治療への応用」
2017年
- 公益財団法人 大和証券ヘルス財団 第44回調査研究助成 2017年 代表「アルツハイマー型認知症のゲノム解析による発症リスク遺伝子の同定と早期予防のための発症予測モデルの開発」
- 文部科学省科学研究費補助金 新学術領域 (研究領域提案型) 2017年-2021年 分担「代謝アダプテーションのトランスオミクス解析」
2016年
- AMED ゲノム医療実現推進プラットフォーム事業 (先端ゲノム研究開発) タイプA 2016年-2020年 分担「糖尿病の遺伝・環境因子の包括的解析から日本発次世代型精密医療を実現するプロジェクト」
- 公益財団法人 金原一郎記念医学医療振興財団 第31回基礎医学医療研究助成金 2016年 代表「エクソームデータからLong INDEL検出法の開発とLong INDELを介した疾患原因遺伝子の解明」
- 平成28年度 学長裁量優秀若手研究者奨励賞 2016年 代表「エクソーム解析によるQT延長症候群の疾患原因変異の網羅的探索」
- 文部科学省科学研究費補助金 若手研究(B) 2016年-2017年 代表「エクソーム解析によるQT延長症候群の疾患原因変異の網羅的探索」
先頭へ戻る
特許
3. アルツハイマー型認知症発症リスク評価のための方法、キット及びデバイス (2020). 出願番号: 特願2020-131832, 出願日: 令和2年8月3日, 出願人: 大森智織, 須藤裕子, 重水大智, 新飯田俊平, 尾崎浩一, 櫻井孝.
2. 海馬萎縮を検出するためのキット又はデバイス及び方法 (2020). 出願番号: 特願2020-64383, 出願日: 令和2年3月31日, 出願人: 大森智織, 須藤 裕子, 吉本真紀子, 新飯田俊平, 伊藤健吾, 重水大智, 加藤隆司, 中村昭範.
1. 認知症の検出のためのキット又はデバイス及び方法 (2018). 出願番号: 特願2018-085652, 出願日: 平成30年4月26日, 出願人: 重水大智, 新飯田俊平, 櫻井孝, 鈴木佳奈, 河内淳平, 須藤裕子, 吉本真紀子, 小園聡子, 近藤哲司.
先頭へ戻る


 Mol Med. 2025 Mar 6;31(1):87. (プレスリリース)
Mol Med. 2025 Mar 6;31(1):87. (プレスリリース) . Sci Rep. 2025 Jan 30;15(1):3838. (News & Topics)
. Sci Rep. 2025 Jan 30;15(1):3838. (News & Topics) . NPJ Aging. 2024 Nov 21;10(1):52. (News & Topics)
. NPJ Aging. 2024 Nov 21;10(1):52. (News & Topics) . Gerontology. 2024;70(6):630-638. (News & Topics)
. Gerontology. 2024;70(6):630-638. (News & Topics) . J Hum Genet. 2024 May;69(5):171-176. (News & Topics)
. J Hum Genet. 2024 May;69(5):171-176. (News & Topics) . NPJ Aging. 2024 Jan 2;10(1):3. (プレスリリース)
. NPJ Aging. 2024 Jan 2;10(1):3. (プレスリリース) . Gerontology. 2024;70(3):279-289.
. Gerontology. 2024;70(3):279-289. Transl Psychiatry. 2023 June 29;13:232.
Transl Psychiatry. 2023 June 29;13:232. J Gerontol A Biol Sci Med Sci. 2023 Jun 22;glad150.
J Gerontol A Biol Sci Med Sci. 2023 Jun 22;glad150. NPJ Aging. 2022 Nov 4;8(1):15.
NPJ Aging. 2022 Nov 4;8(1):15. Am J Med Genet B Neuropsychiatr Genet. 2022 Jun 28.
Am J Med Genet B Neuropsychiatr Genet. 2022 Jun 28. Mol Psychiatry. 2022 Mar 10.
Mol Psychiatry. 2022 Mar 10. J Hum Genet. 2021 Nov 5.
J Hum Genet. 2021 Nov 5. Database (Oxford) 2021 Nov 3;2021(2021):baab072.
Database (Oxford) 2021 Nov 3;2021(2021):baab072. Sci Rep. 2021 Oct 22;11(1):20947.
Sci Rep. 2021 Oct 22;11(1):20947. Genome Med. 2021 Apr 29;13(1):65.
Genome Med. 2021 Apr 29;13(1):65. Transl Psychiatry.2021 Mar 3;11(1):151.
Transl Psychiatry.2021 Mar 3;11(1):151. Alzheimers Res Ther. 2020 Nov 10;12(1):145.
Alzheimers Res Ther. 2020 Nov 10;12(1):145. PLoS One. 2020 Sep 29;15(9):e0239196.
PLoS One. 2020 Sep 29;15(9):e0239196. Alzheimers Res Ther. 2020 Jul 16;12(1):87.
Alzheimers Res Ther. 2020 Jul 16;12(1):87. BMC Mol Cell Biol. 2019 Dec 20;20(Suppl 2):57.
BMC Mol Cell Biol. 2019 Dec 20;20(Suppl 2):57. BMC Med Genomics. 2019 Oct 30;12(1):150.
BMC Med Genomics. 2019 Oct 30;12(1):150. Sci Rep. 2019 Aug 6;9(1):11399.
Sci Rep. 2019 Aug 6;9(1):11399. Genome Med. 2019 Jul 24;11(1):44.
Genome Med. 2019 Jul 24;11(1):44. Mol Med. 2019 Jun 20;25(1):2.
Mol Med. 2019 Jun 20;25(1):2. Commun Biol. 2019 Feb.25;2:77.
Commun Biol. 2019 Feb.25;2:77. BMC Bioinformatics. 2019 19(Suppl 13):547.
BMC Bioinformatics. 2019 19(Suppl 13):547. Hum Genet. 2018 Jul. 137(6-7):521-533.
Hum Genet. 2018 Jul. 137(6-7):521-533. Encyclopedia of Bioinformatics and Computational Biology 2018 Sep 1, 20089;1-24.
Encyclopedia of Bioinformatics and Computational Biology 2018 Sep 1, 20089;1-24. Front Genet. 2018 Apr 24, 9:115.
Front Genet. 2018 Apr 24, 9:115. Sci Rep. 2018 Apr 4;8(1):5608.
Sci Rep. 2018 Apr 4;8(1):5608. Cancer Med. 2017 Jul;6(7):1627-1638.
Cancer Med. 2017 Jul;6(7):1627-1638. IEEE Trans Biomed Eng. 2017 Jan;64(1):112-122.
IEEE Trans Biomed Eng. 2017 Jan;64(1):112-122. Circ J. 2016 Nov 25;80(12):2435-2442.
Circ J. 2016 Nov 25;80(12):2435-2442. BMC Bioinformatics. 2016 Aug 24;17(1):319.
BMC Bioinformatics. 2016 Aug 24;17(1):319. J Am Heart Assoc. 2016 Sep 13;5(9):e003644.
J Am Heart Assoc. 2016 Sep 13;5(9):e003644. Oncotarget. 2016 Mar 1;7(9):10182-92.
Oncotarget. 2016 Mar 1;7(9):10182-92. Circulation. 2015 132(Suppl 3) A12257.
Circulation. 2015 132(Suppl 3) A12257. Sci Rep. 2015 Aug 3;5:12742.
Sci Rep. 2015 Aug 3;5:12742. PLoS One. 2015 10(7), e0130329.
PLoS One. 2015 10(7), e0130329. Sci Rep. 2015 Mar 19;5:9331.
Sci Rep. 2015 Mar 19;5:9331. Nat Commun. 2015 Jan 30;6:6120.
Nat Commun. 2015 Jan 30;6:6120. Eur J Hum Genet. 2014 Mar;23(3):374-380.
Eur J Hum Genet. 2014 Mar;23(3):374-380. Circulation. 2014 130(Suppl 2) A13817.
Circulation. 2014 130(Suppl 2) A13817. Circ Cardiovasc Genet. 2014 Aug;7(4):466-474.
Circ Cardiovasc Genet. 2014 Aug;7(4):466-474. PLoS One. 2014 9(3), e92549.
PLoS One. 2014 9(3), e92549. Circulation. 2013 128(22) A13371.
Circulation. 2013 128(22) A13371. J Clin Endocrinol Metab. 2013 98(10), E1667-E1673.
J Clin Endocrinol Metab. 2013 98(10), E1667-E1673. Sci Rep. 2013 3:2161. (natureasia.com注目の論文
Sci Rep. 2013 3:2161. (natureasia.com注目の論文 )
) Hum Mol Genet. 2012 21(9), 2076-2085.
Hum Mol Genet. 2012 21(9), 2076-2085. Clin Cancer Res. 2012 18(5), 1374-1385.
Clin Cancer Res. 2012 18(5), 1374-1385. PLoS Comput Biol. 2012 8(2), e1002347.
PLoS Comput Biol. 2012 8(2), e1002347. J Chem Inf Model. 2011 51(11), 2977-2985.
J Chem Inf Model. 2011 51(11), 2977-2985. Nucleic Acids Res. 2010 38(suppl 2), W138-W143.
Nucleic Acids Res. 2010 38(suppl 2), W138-W143. Genome Inform.2010 22, 167-175.
Genome Inform.2010 22, 167-175. J Chem Inf Model. 2009 49(4), 1122-1129.
J Chem Inf Model. 2009 49(4), 1122-1129. Algorithms in Bioinformatics Lecture Notes in Computer Science. 2004 Vol. 3240,147-158.
Algorithms in Bioinformatics Lecture Notes in Computer Science. 2004 Vol. 3240,147-158.